Презентація на тему:
4_10__3_Регуляція транскрипції еукаріот 4 10 11
Завантажити презентацію
4_10__3_Регуляція транскрипції еукаріот 4 10 11
Завантажити презентаціюПрезентація по слайдам:
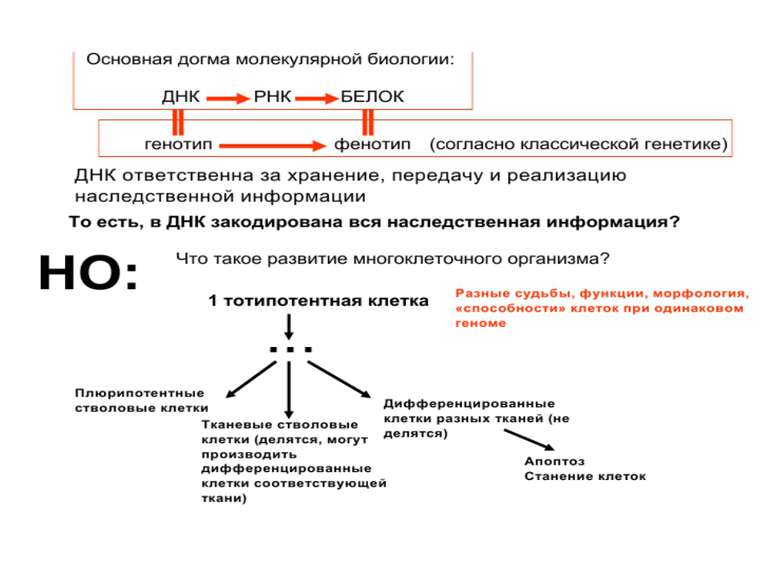
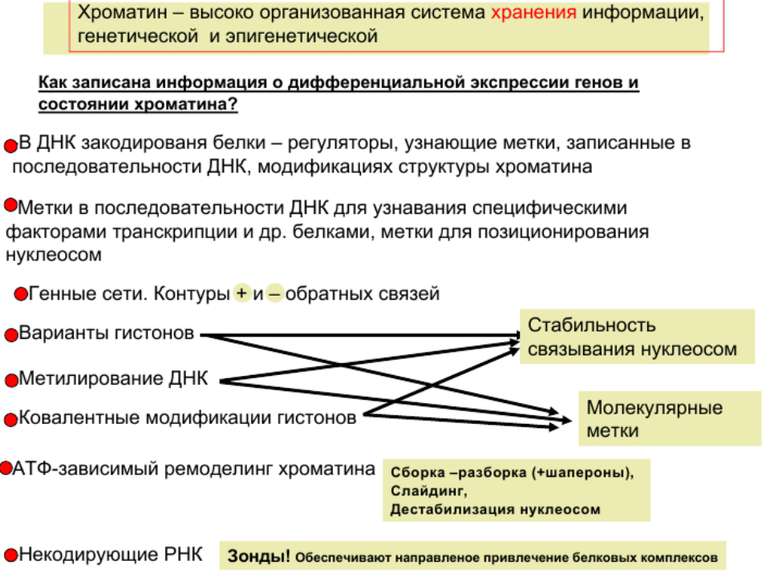
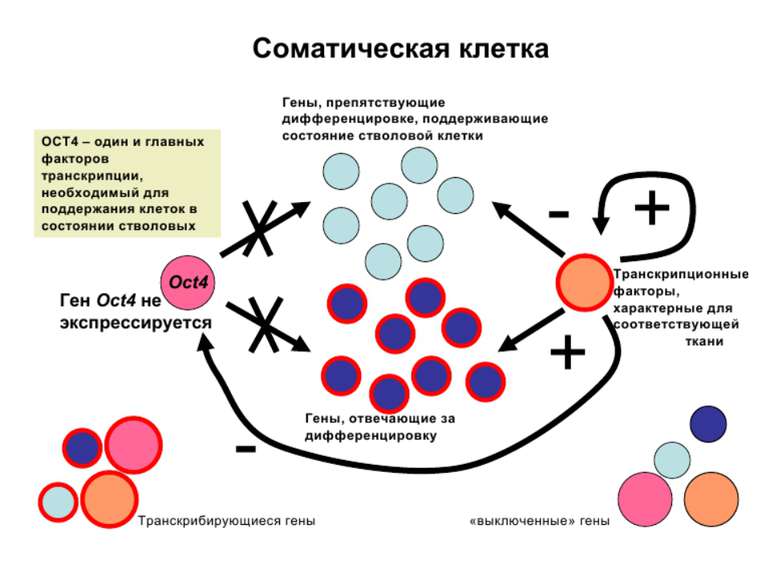
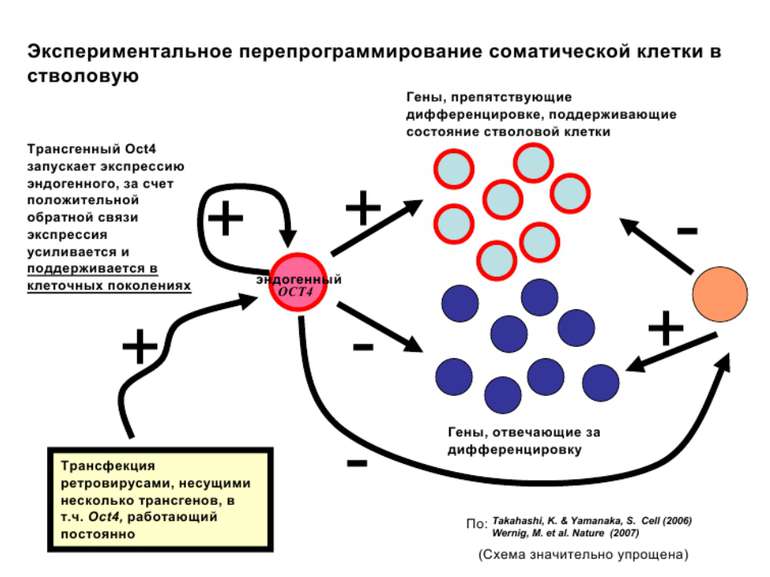
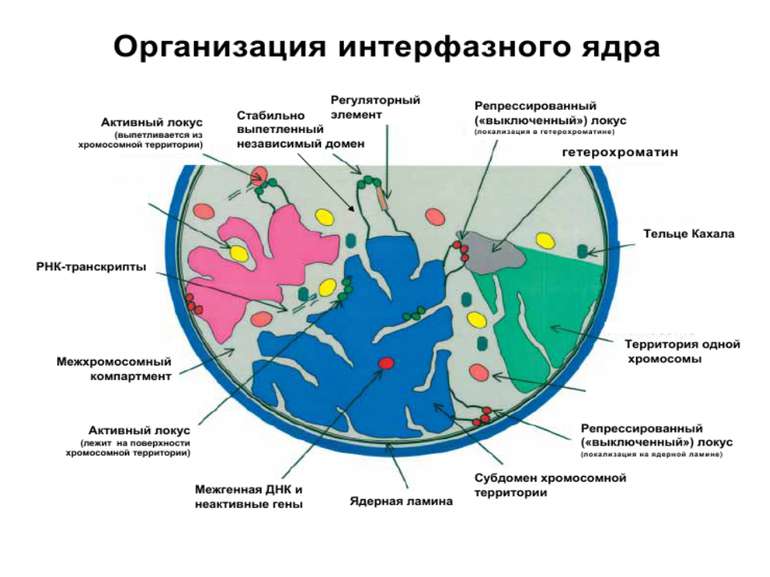
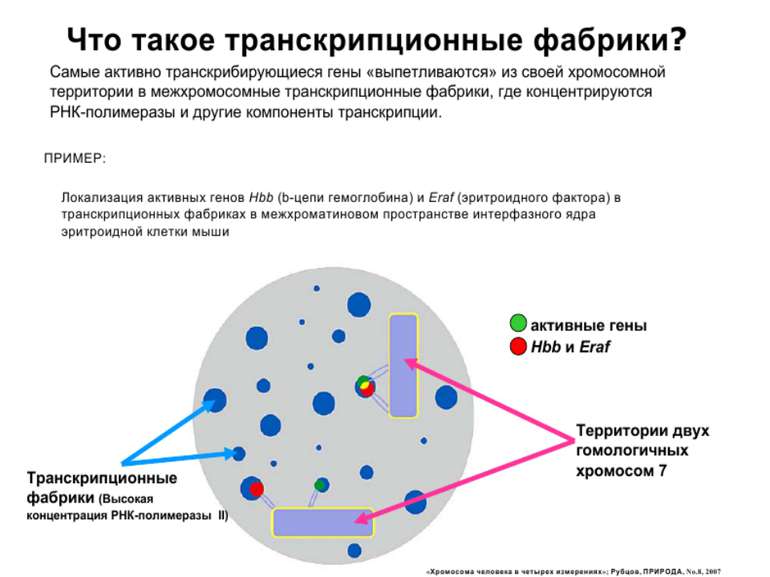
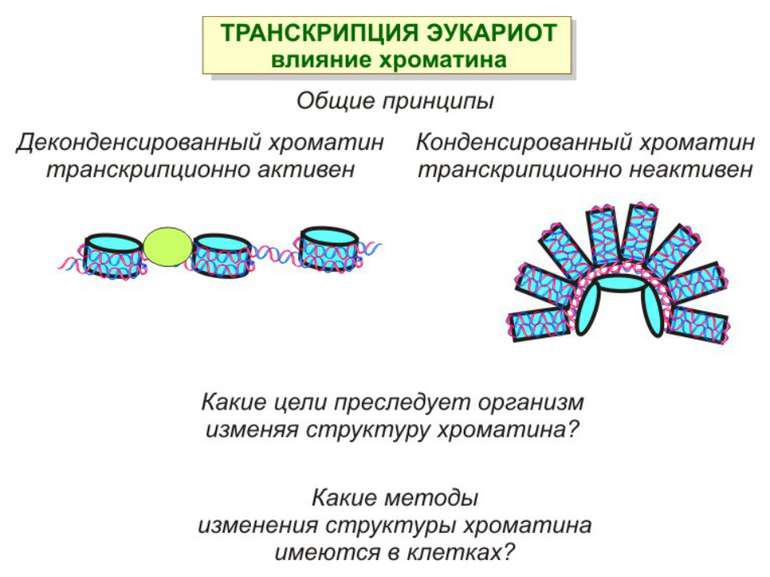
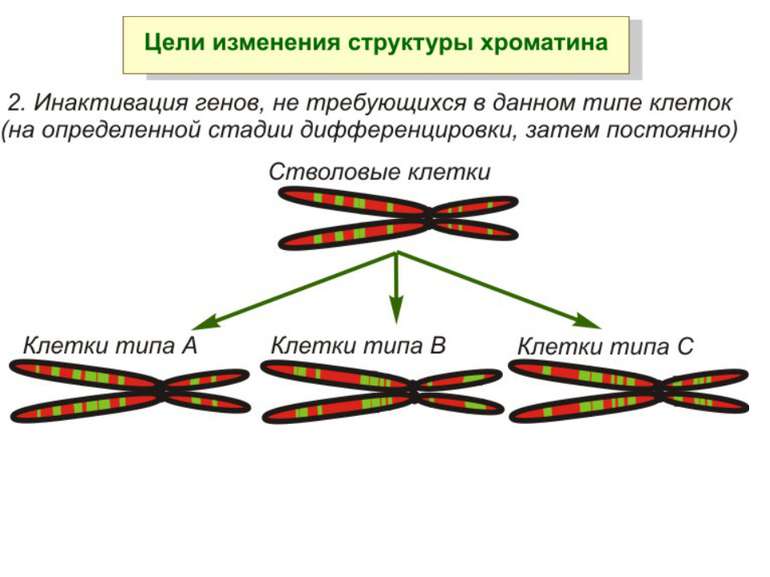
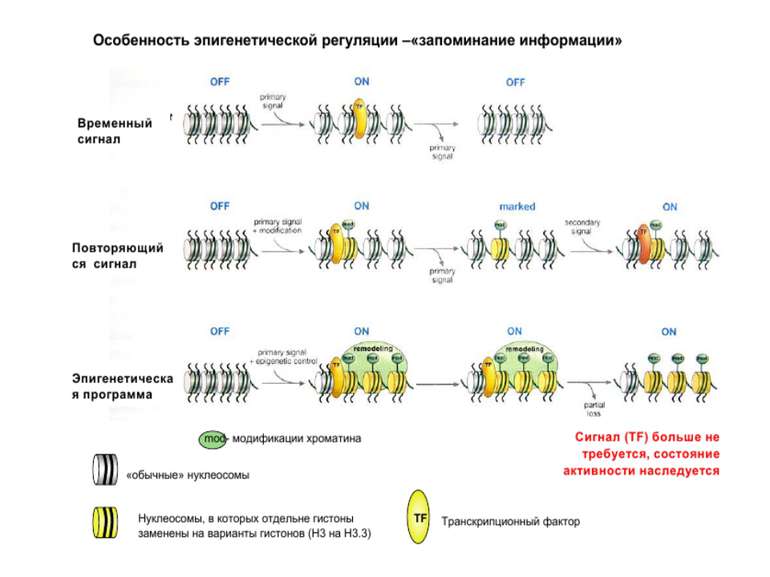
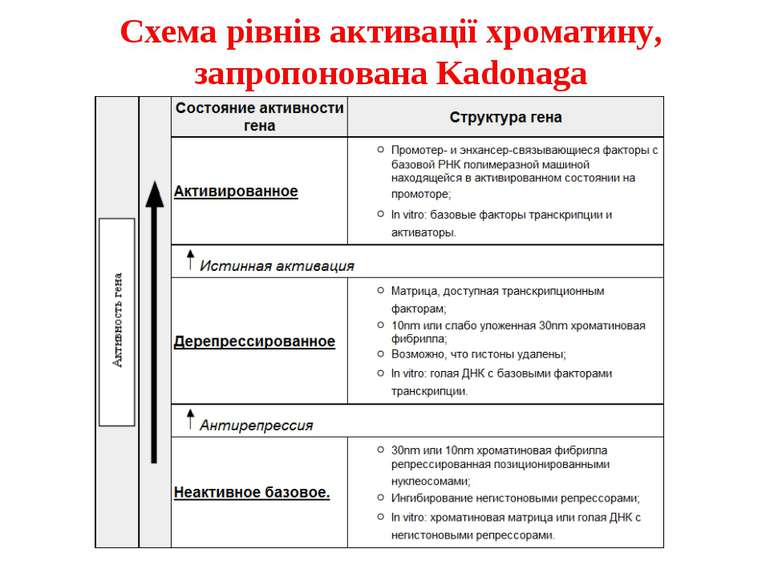
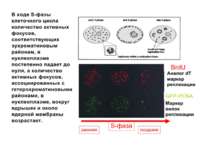
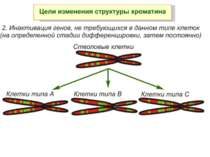
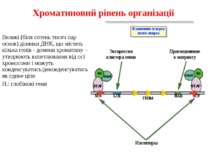
Регуляція експресії генів може здійснюватись на різних рівнях Хроматиновий рівень – конденсація хроматину та доменів хроматину Численні ділянки геному регулюються як єдині протяжні домени хроматину. На цьому рівні можлива епігенетична регуляція (епігенетичне успадкування стану експресії) Окремі гени У цій регуляцї беруть участь: Ковалентні модифікації ДНК Варіанти і модифікації гістонів Різні негістонові білки хроматину Просторова організація ядра Просторово-часова картина реплікації
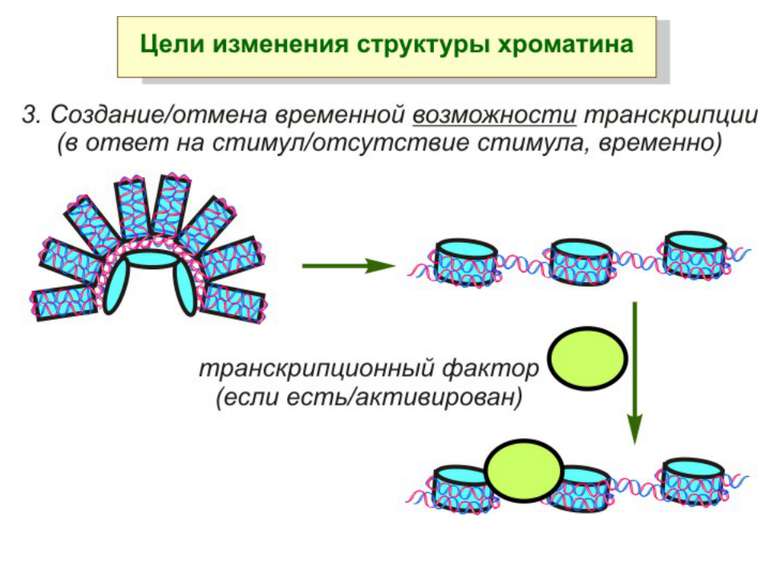
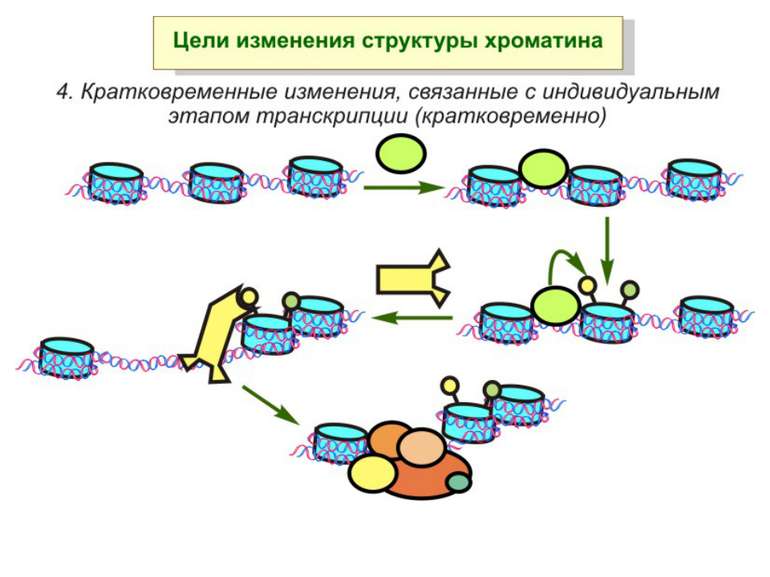
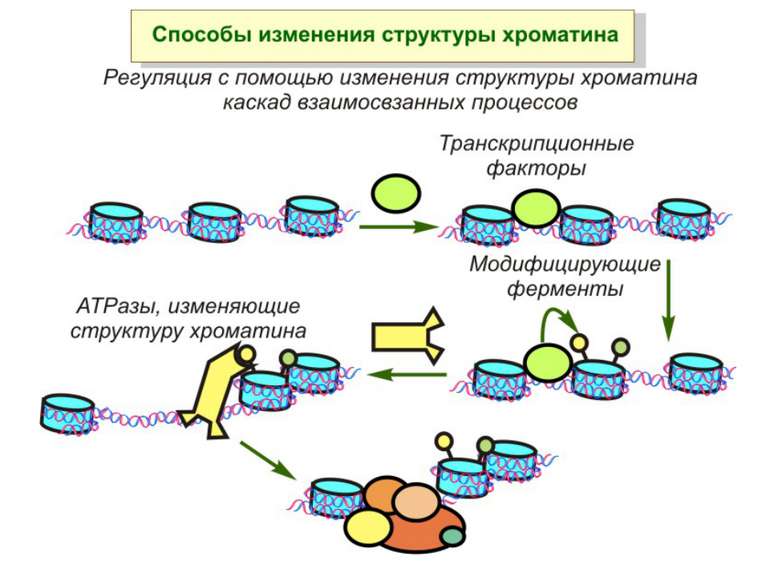
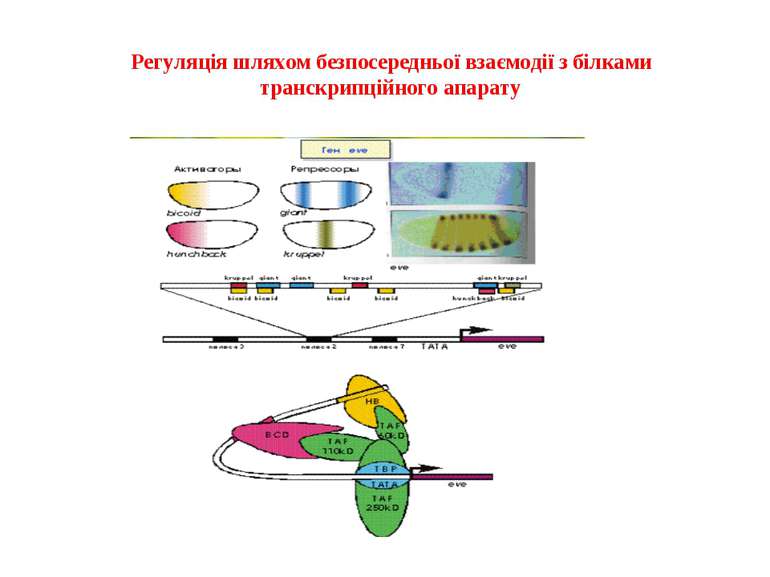
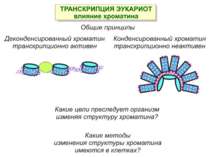
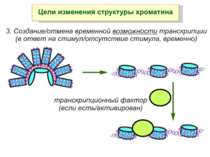
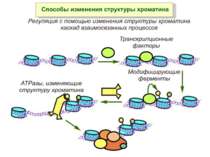
Механізми регуляції транскрипції еукаріот Зв’язування з ДНК регуляторних білків і факторів транскрипції. Модифікація ДНК (метилювання) і гістонів (ацетилювання, метилювання і ін.) Компактизація/декомпактизація хроматину, що здійснюється білковими комплексами
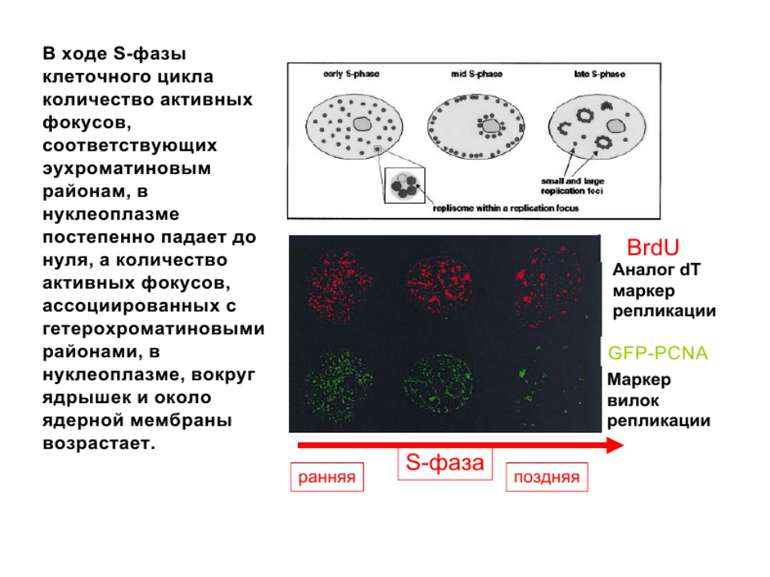
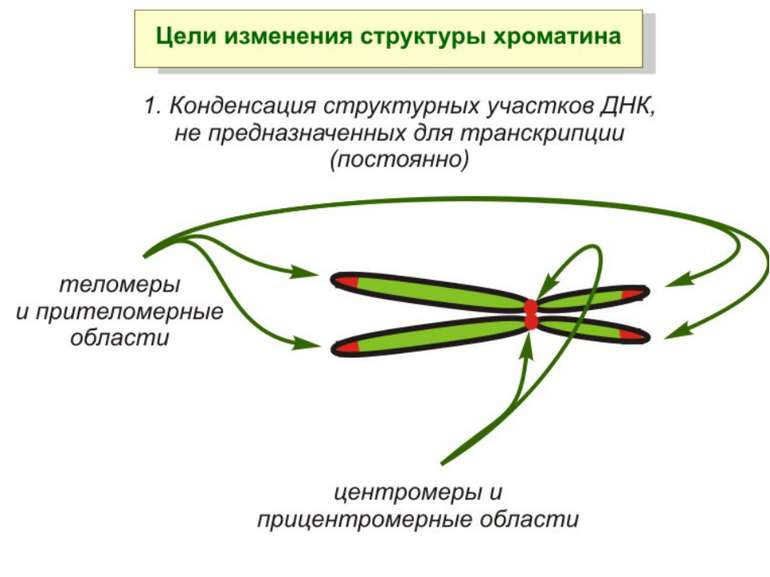
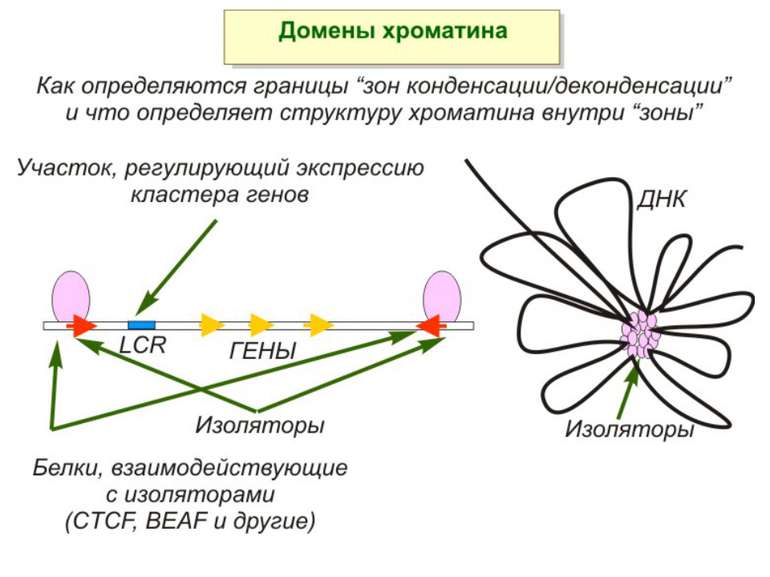
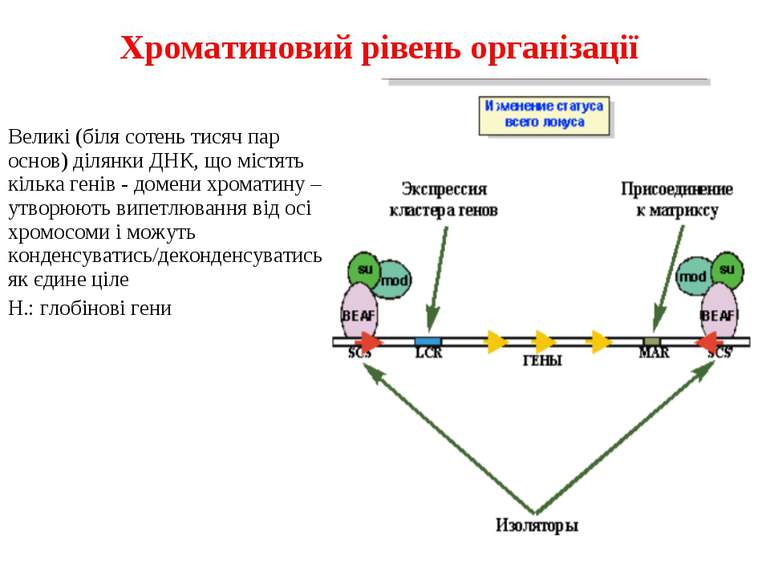
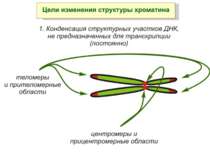
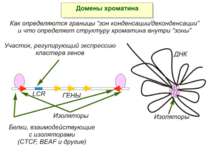
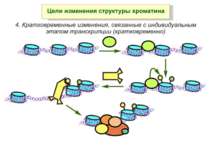
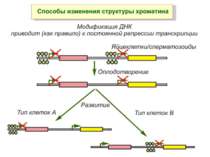
Хроматиновий рівень організації Великі (біля сотень тисяч пар основ) ділянки ДНК, що містять кілька генів - домени хроматину – утворюють випетлювання від осі хромосоми і можуть конденсуватись/деконденсуватись як єдине ціле Н.: глобінові гени
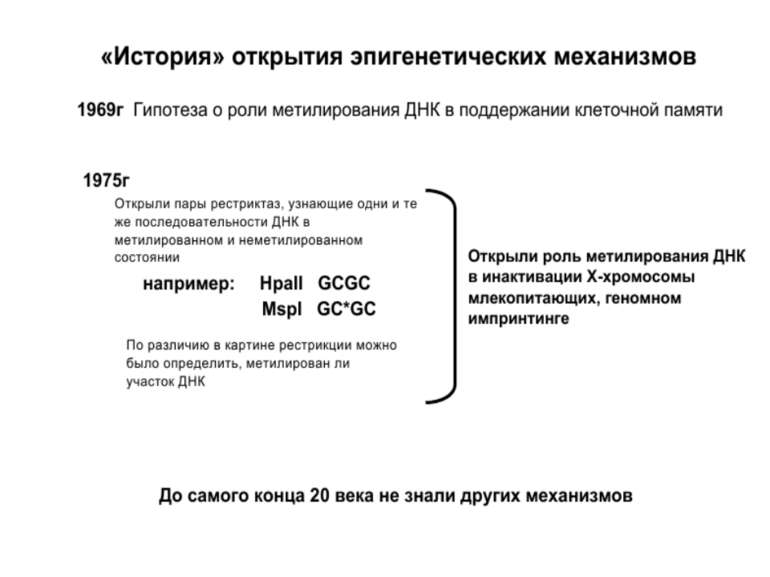
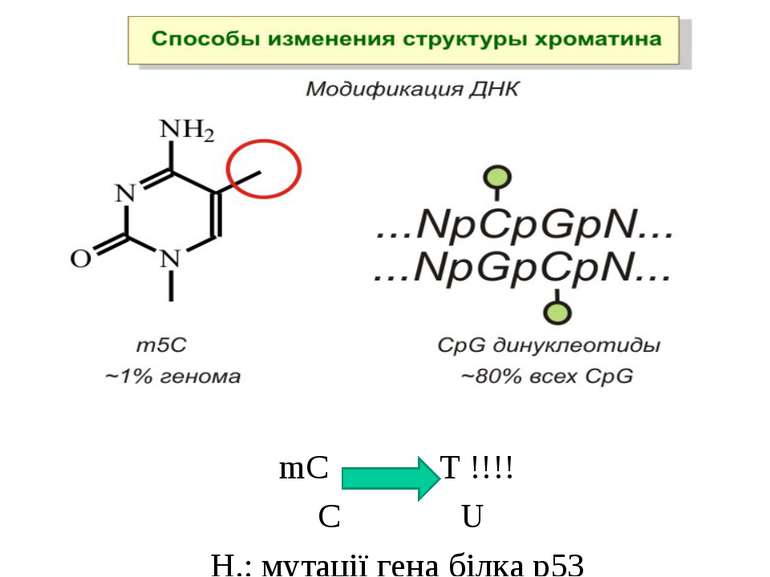
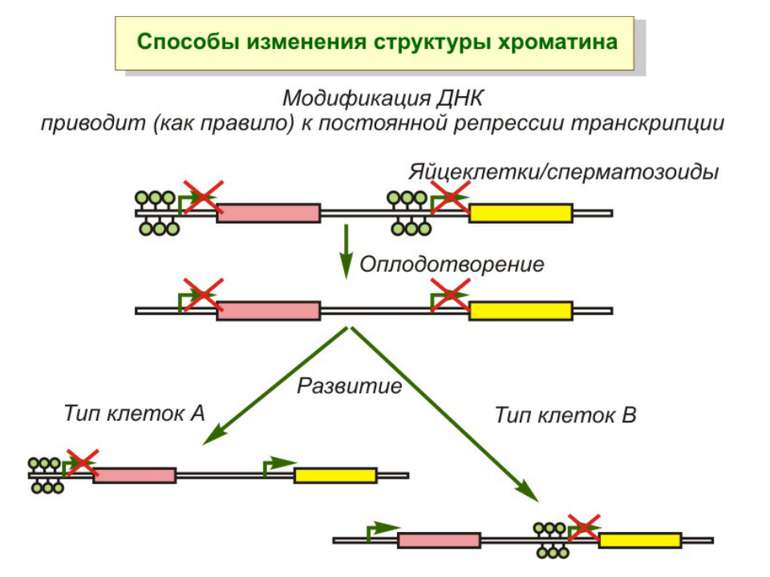
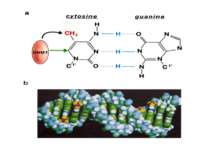
Роль метилювання ДНК в регуляції транскрипції Вибірковість метилювання Карти метилювання (батьківський імпрінтинг, хвилі онтогенезу) Метилювання і онкогенез Участь в інактивації Х-хромосоми (синдром Тернера)
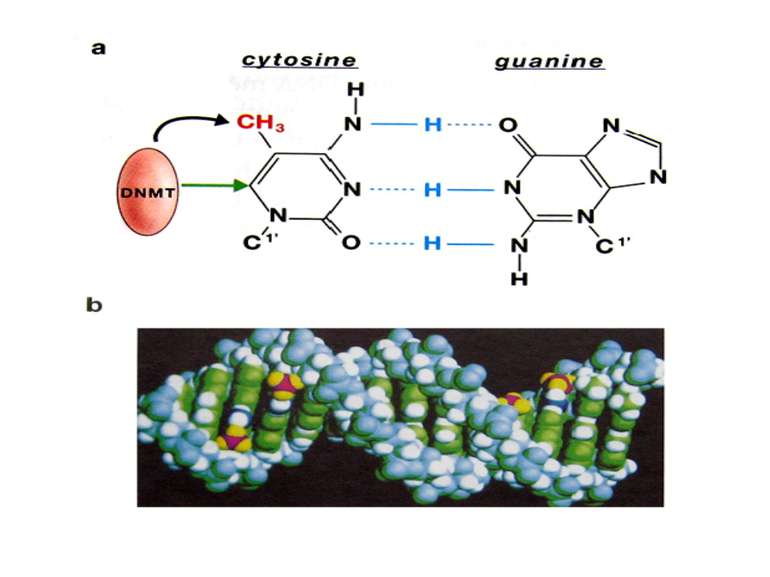
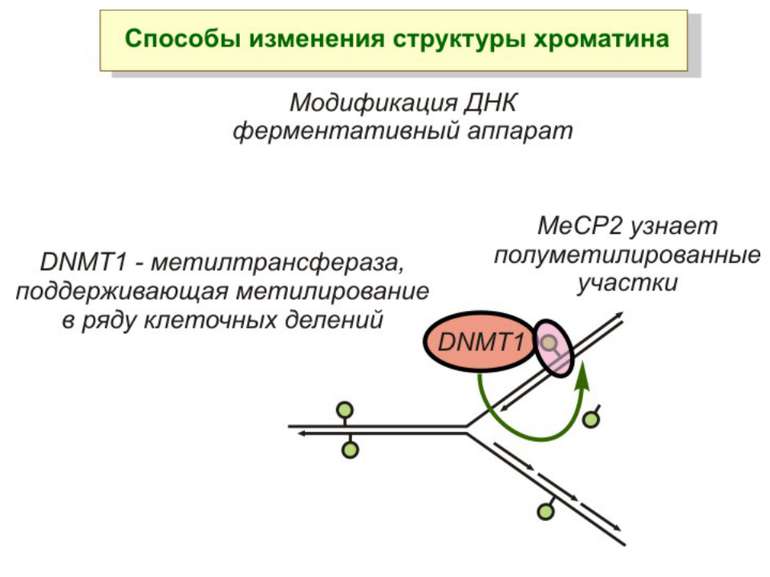
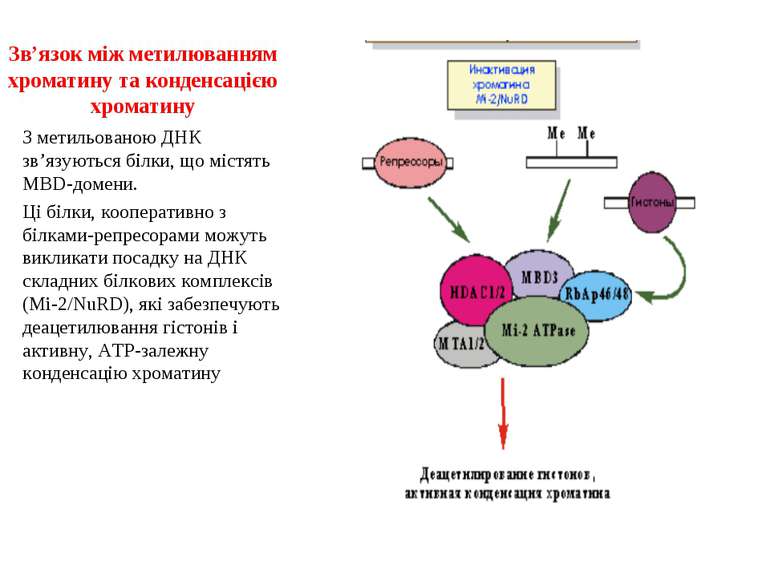
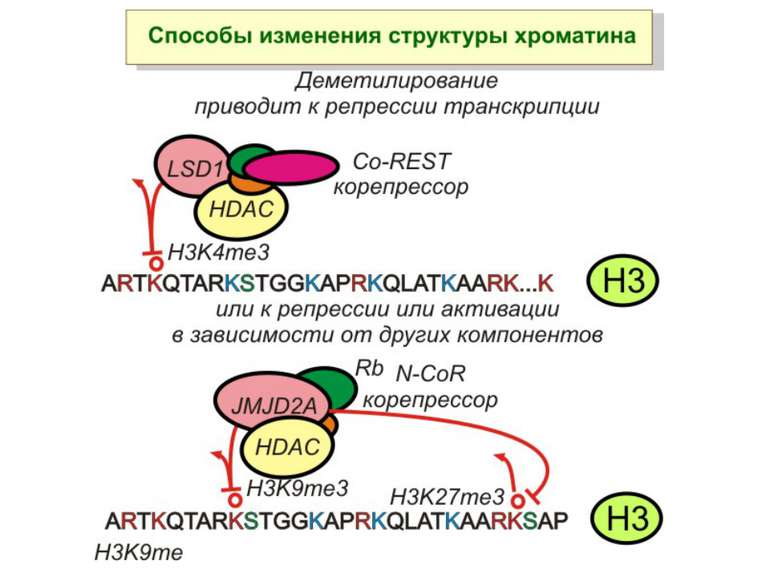
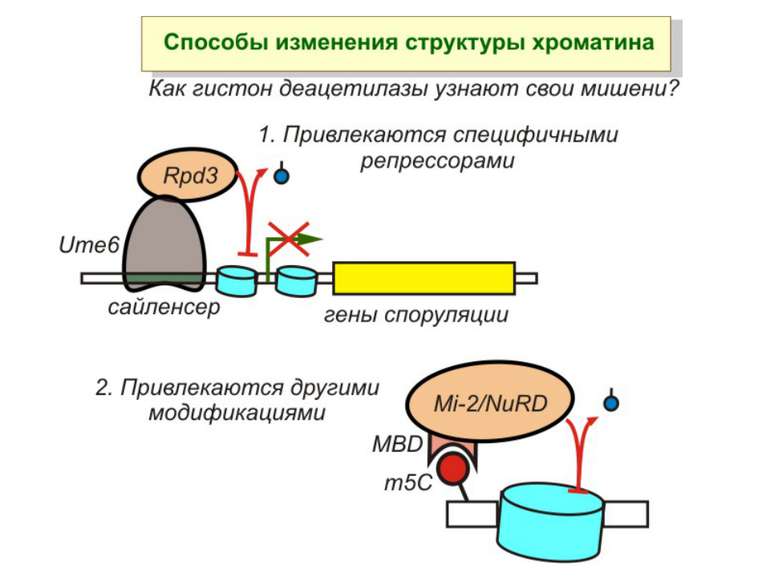
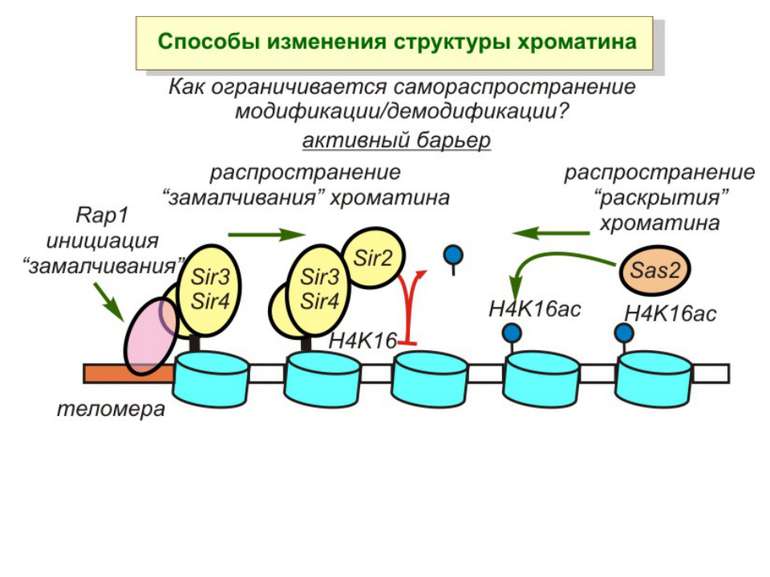
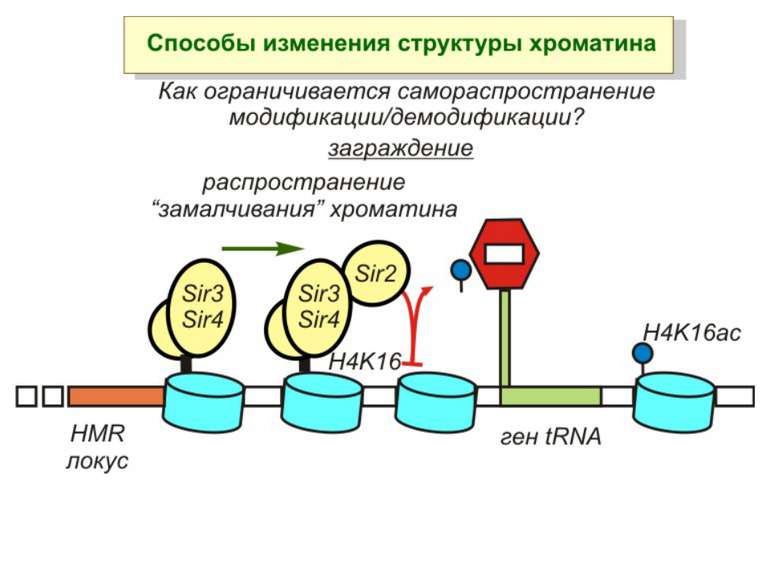
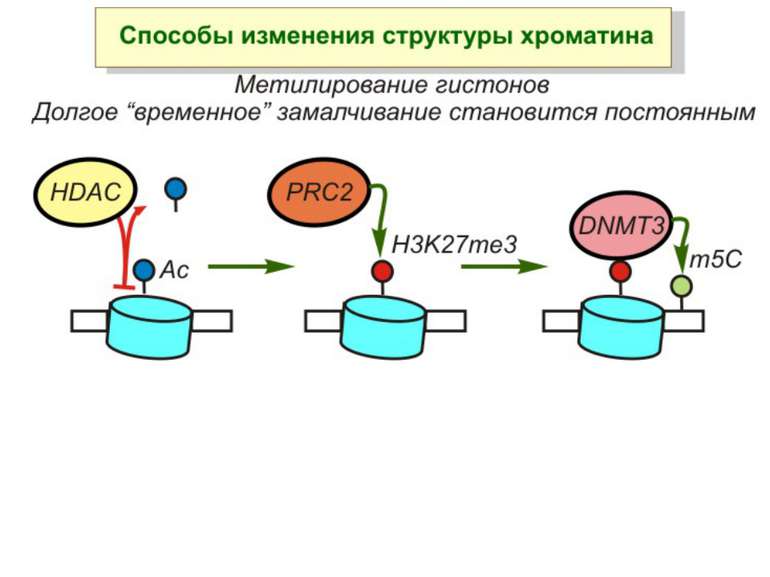
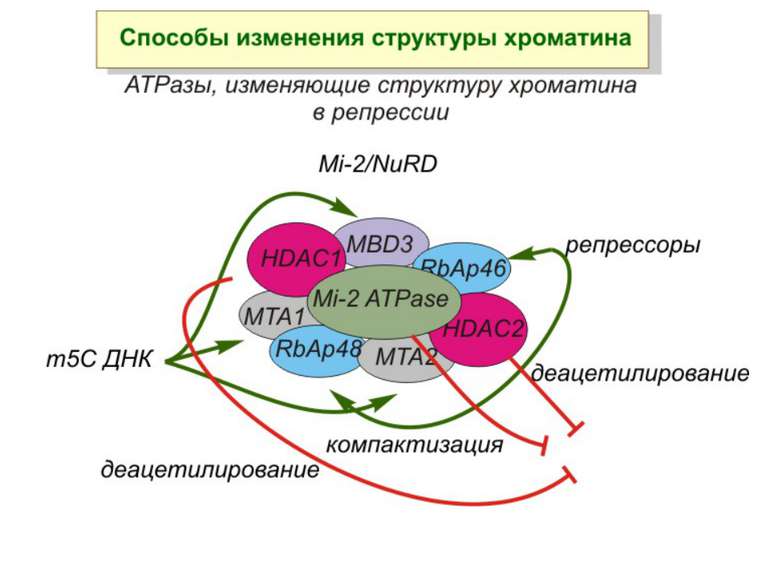
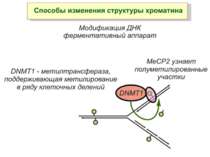
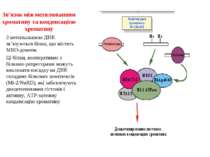
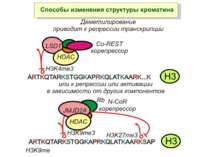
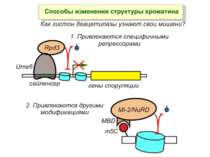
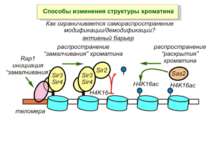
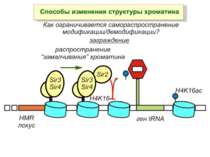
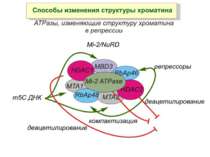
Зв’язок між метилюванням хроматину та конденсацією хроматину З метильованою ДНК зв’язуються білки, що містять MBD-домени. Ці білки, кооперативно з білками-репресорами можуть викликати посадку на ДНК складних білкових комплексів (Mi-2/NuRD), які забезпечують деацетилювання гістонів і активну, ATP-залежну конденсацію хроматину
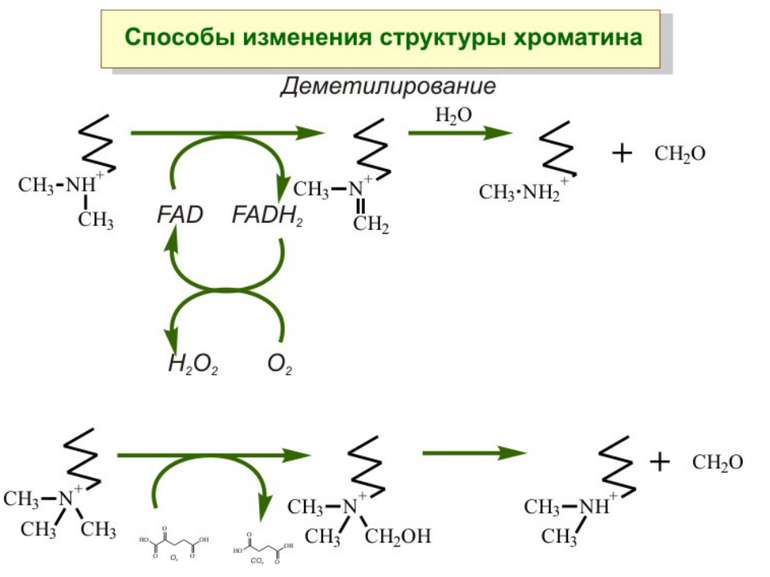
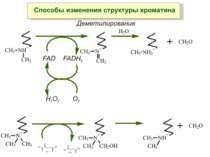
Передача карт метилювання Хвилі метилювання в онтогенезі (пасивне і активне деметилювання ((фактори транскрипції?))
Передача карт метилювання Батьківський імпритинг – сімейні спадкові захворювання аА* і аа Характерне для хребетних
Передача карт метилювання Еволюційне значення “Конфлікт інтересів” Ген фактору росту плаценти активний, якщо успадковується від батька, і гіперметильований (неактивний), якщо приходить від матері. Навпаки, ген, що інгібує фактор росту, активний лише в тому випадку, якщо успадковується від матері.
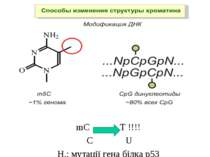
Метилювання і онкогенез Метилювання промоторів генів-супресорів Н.: р53 Rb (retinoblastoma)
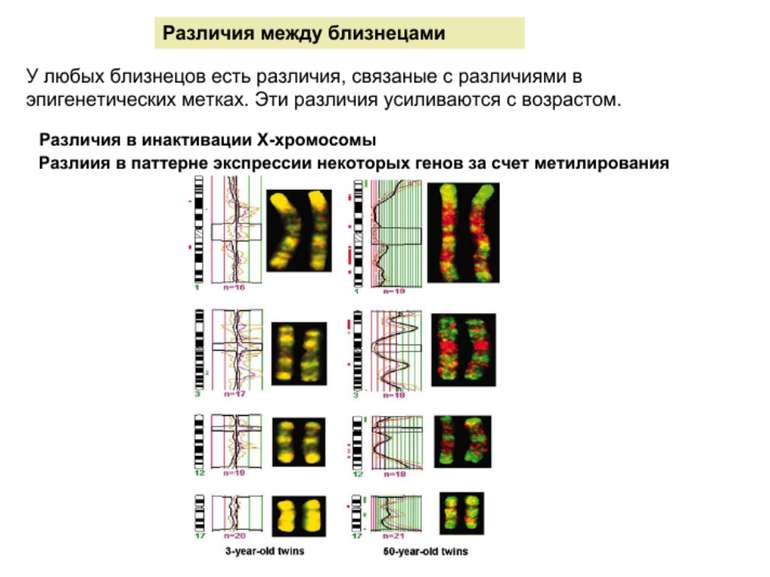
Метилювання і інактивація Х-хромосоми Генний баланс – недопустимість дуплікацій аутосом Неповне виключення Випадковий вибір синдром Тернера – вплив походження Х-хромосоми
Метилювання аденінових залишків ДНК N6-метиладенін Залучене в регуляцію експресії генів і реплікацію, в тому числі і мітохондріальних ДНК. Може контролювати персистенцію чужерідних ДНК в клітині і, ймовірно, служить елементом системи рестрикції-модифікації у рослин.
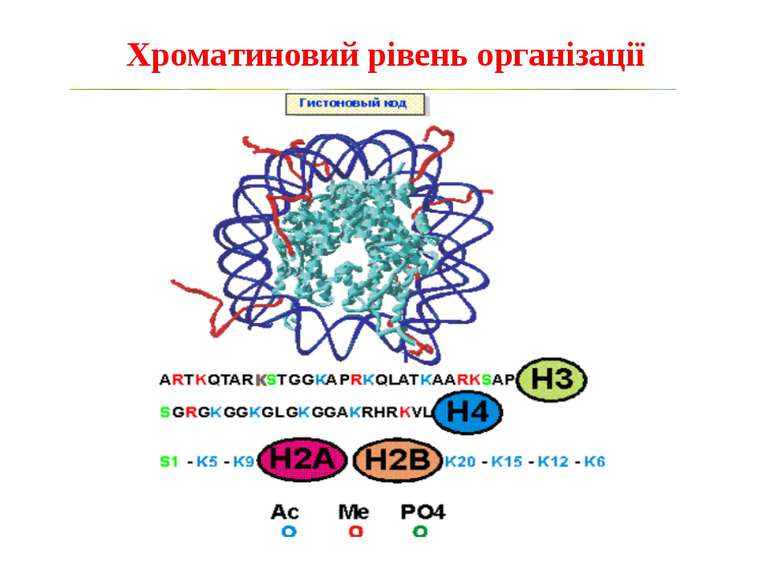
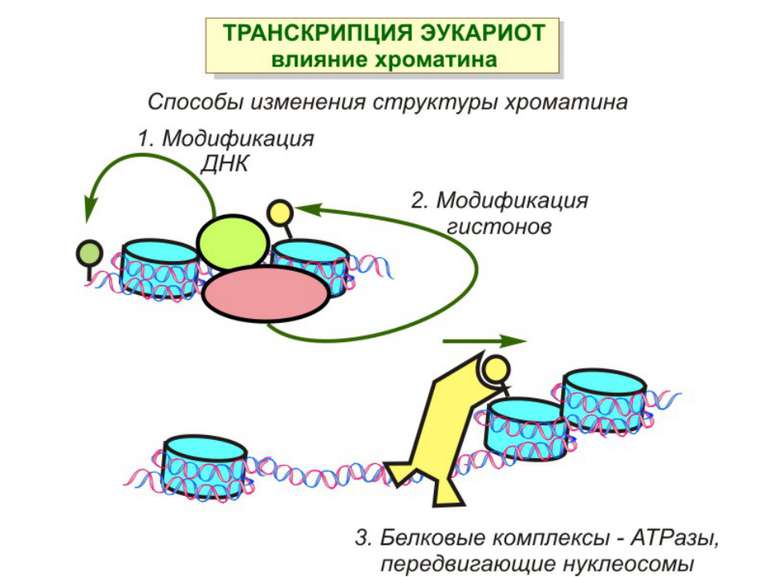
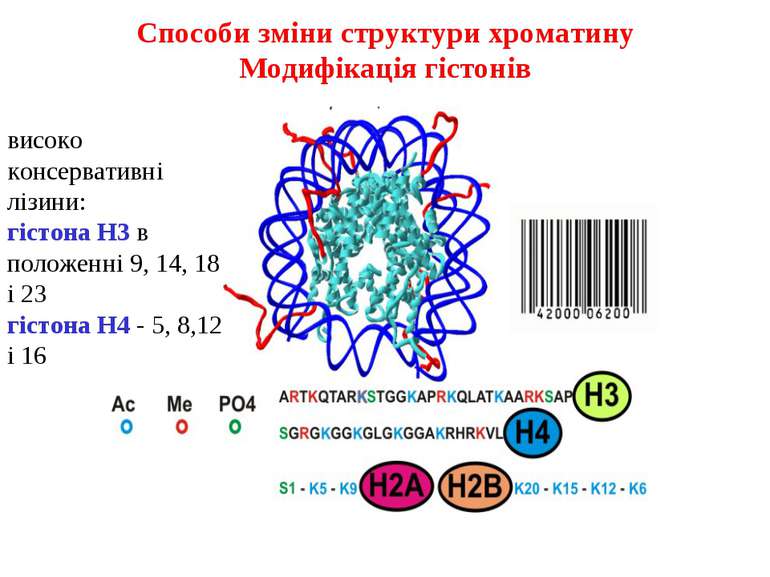
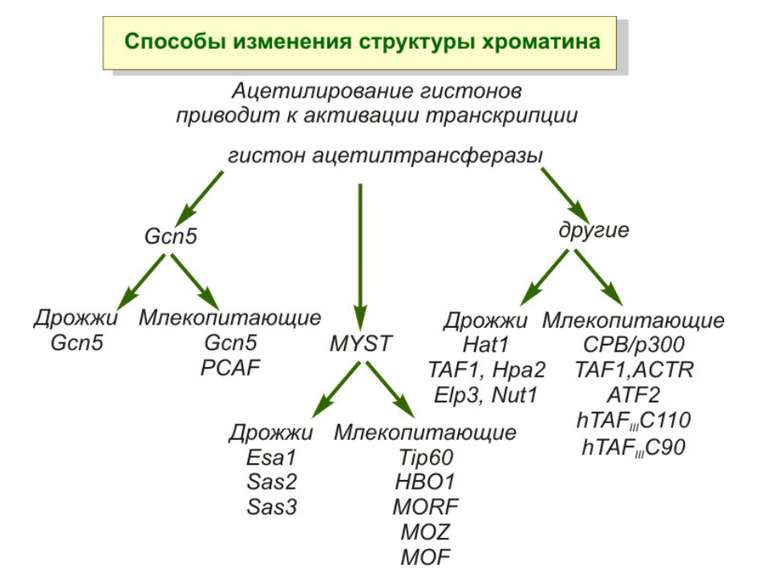
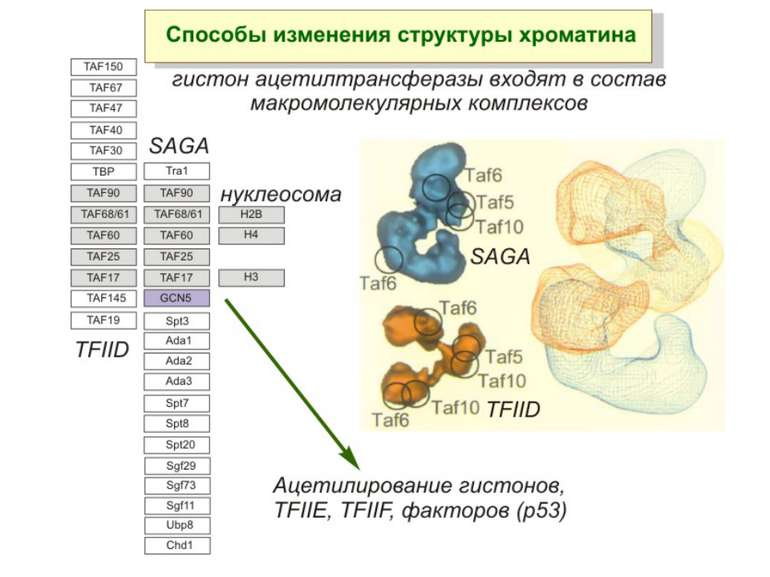
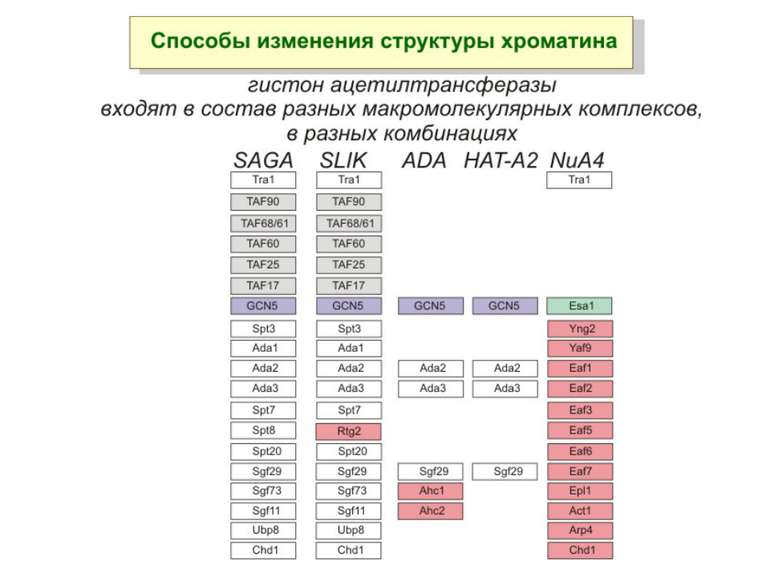
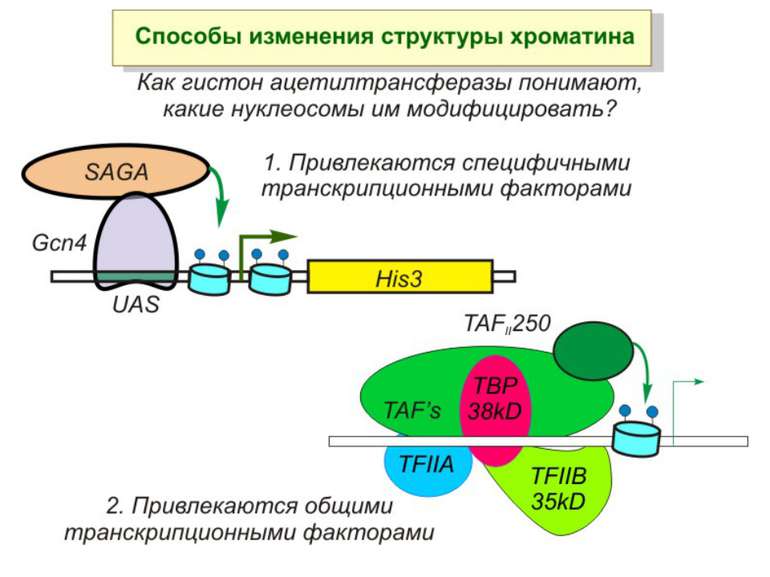
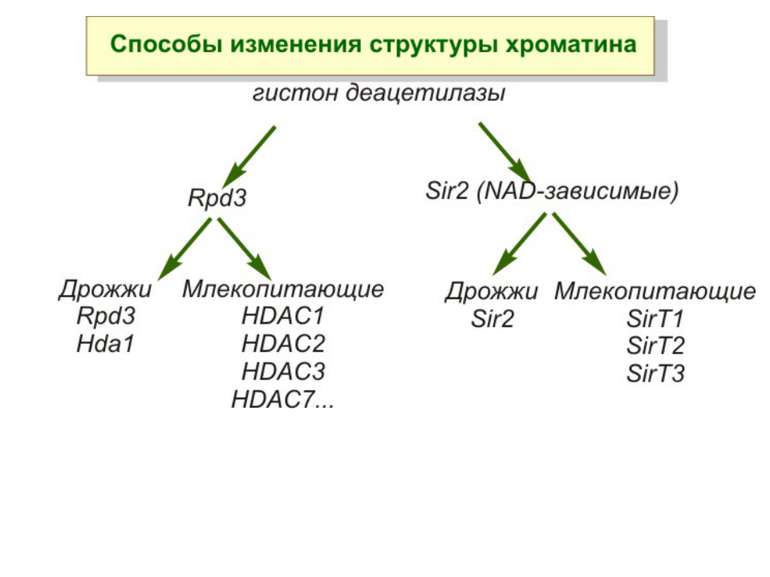
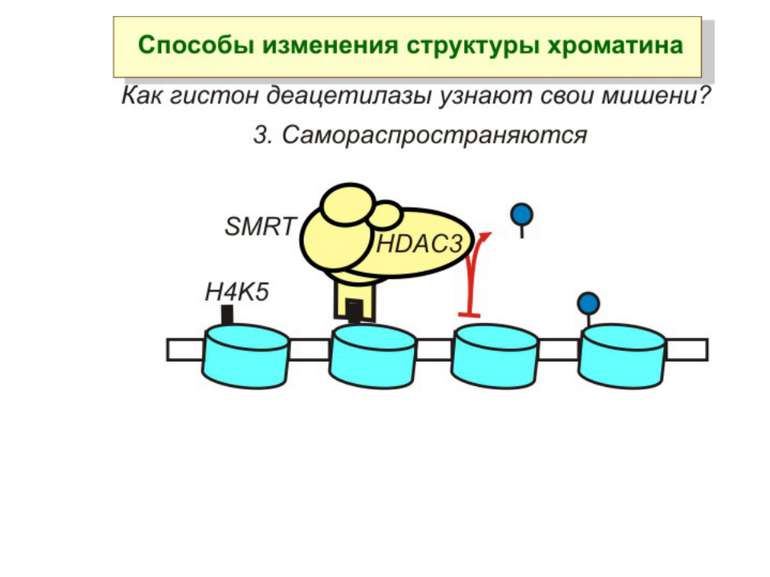
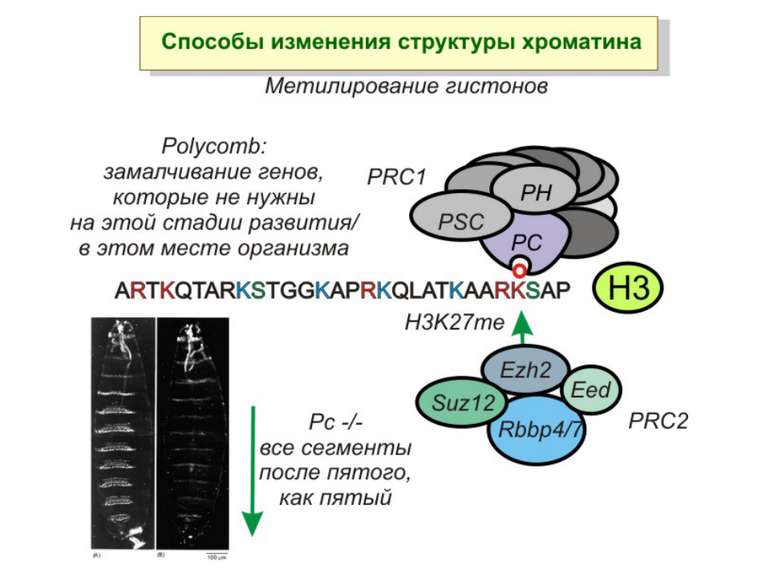
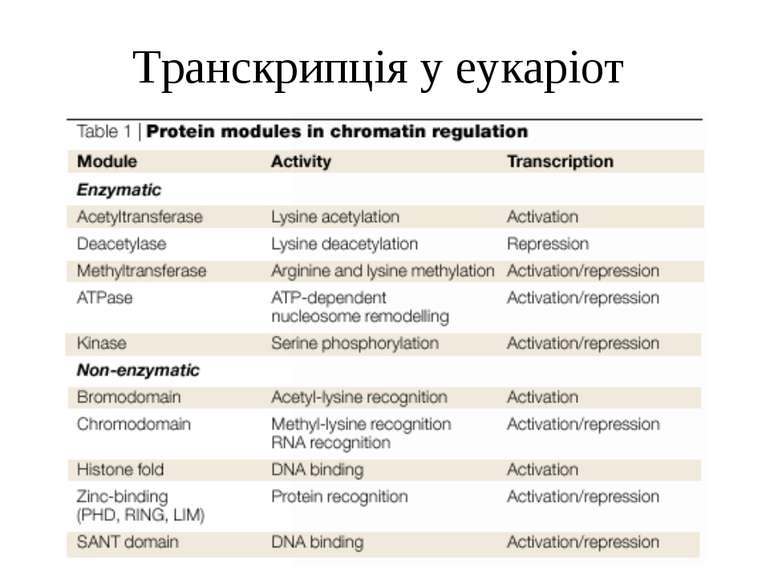
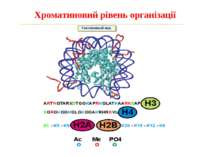
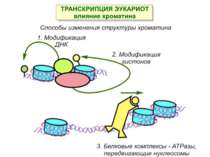
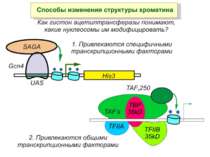
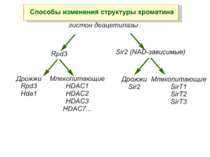
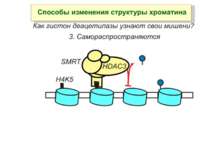
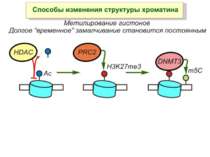
Способи зміни структури хроматину Модифікація гістонів високо консервативні лізини: гістона Н3 в положенні 9, 14, 18 і 23 гістона Н4 - 5, 8,12 і 16
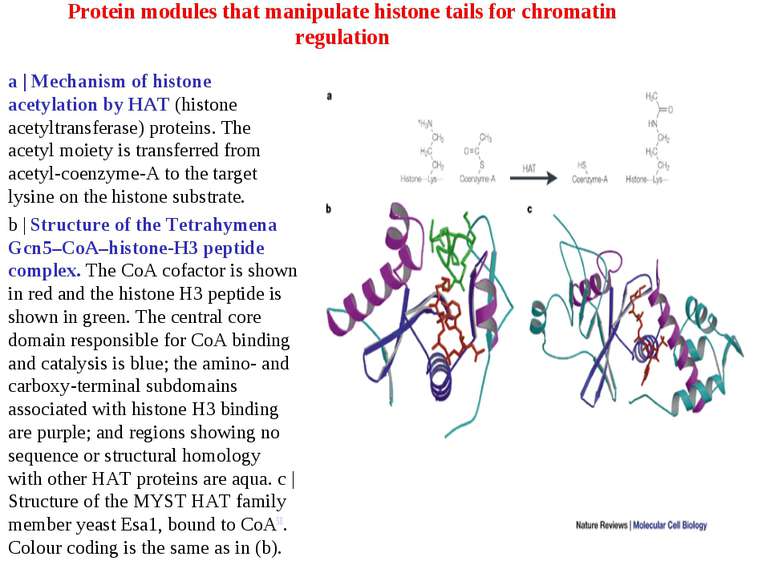
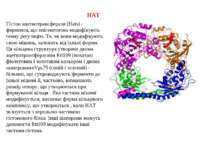
Protein modules that manipulate histone tails for chromatin regulation a | Mechanism of histone acetylation by HAT (histone acetyltransferase) proteins. The acetyl moiety is transferred from acetyl-coenzyme-A to the target lysine on the histone substrate. b | Structure of the Tetrahymena Gcn5–CoA–histone-H3 peptide complex. The CoA cofactor is shown in red and the histone H3 peptide is shown in green. The central core domain responsible for CoA binding and catalysis is blue; the amino- and carboxy-terminal subdomains associated with histone H3 binding are purple; and regions showing no sequence or structural homology with other HAT proteins are aqua. c | Structure of the MYST HAT family member yeast Esa1, bound to CoA58. Colour coding is the same as in (b).
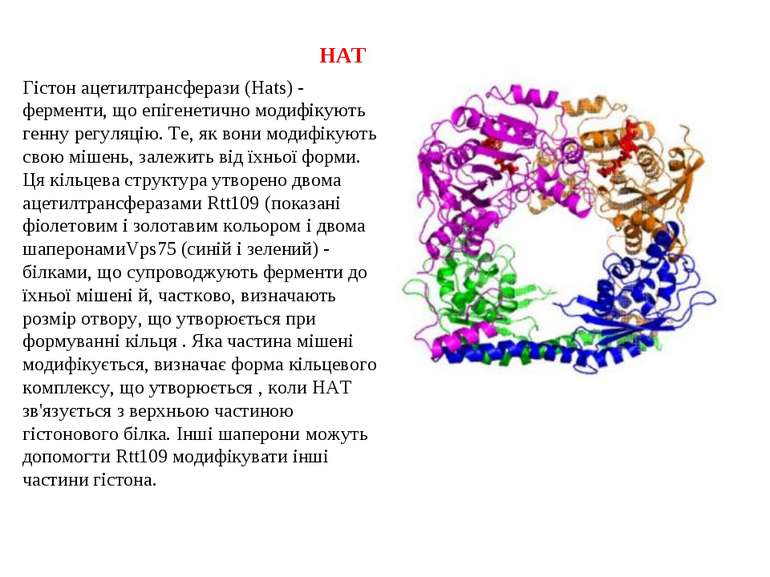
HAT Гістон ацетилтрансферази (Hats) - ферменти, що епігенетично модифікують генну регуляцію. Те, як вони модифікують свою мішень, залежить від їхньої форми. Ця кільцева структура утворено двома ацетилтрансферазами Rtt109 (показані фіолетовим і золотавим кольором і двома шаперонамиVps75 (синій і зелений) - білками, що супроводжують ферменти до їхньої мішені й, частково, визначають розмір отвору, що утворюється при формуванні кільця . Яка частина мішені модифікується, визначає форма кільцевого комплексу, що утворюється , коли HAT зв'язується з верхньою частиною гістонового білка. Інші шаперони можуть допомогти Rtt109 модифікувати інші частини гістона.
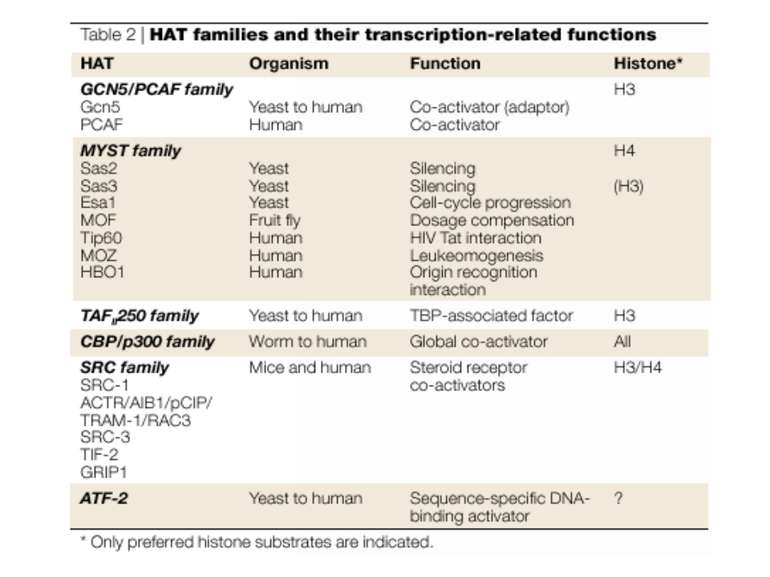
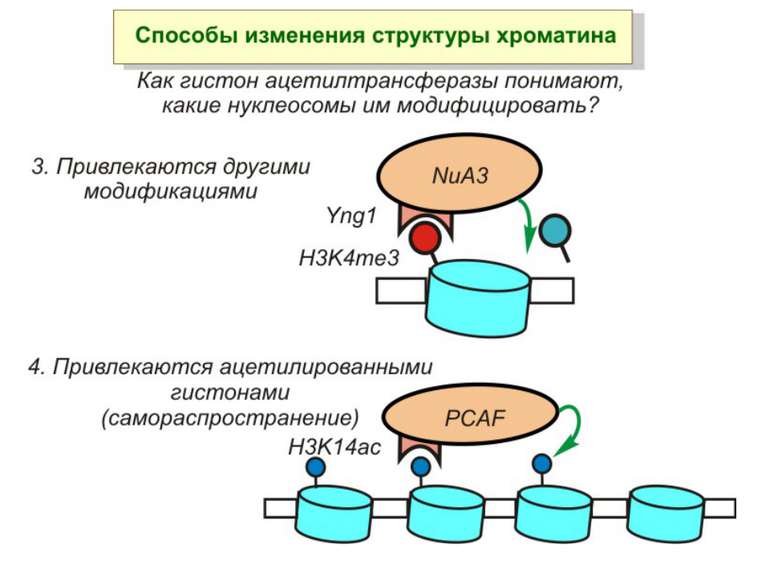
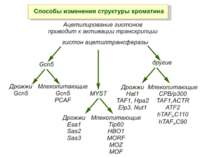
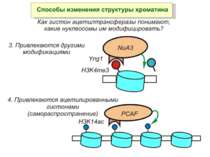
GNAT родина (Gcn5-related N-acetytransferases) включає білки, що беруть участь або зв’язані з ініціацією транскипції (Gcn5 і PCAF), елонгацією (Elp3), депозицією гістонів і теломерним мовчанням (Hat1)
Родина p300/CBP HAT Наявні спільні гомологічні послідовності з GNATs. HAT активність p300 і CBP необхідна для їх роли в трансактивації, вони асоціюють з ін. ацетилтрансферазами. P300/CBP - HAC, взаємодіють з енхансерами і асоціюють з РНК - полімеразою II
Родина MYST Включає членів MOZ (онкоген людини), Ybf2/Sas3, Sas2 і Tip60. Н.: гостра мієлома Участь в транскрипційній елонгації і реплікації та в мовчанні дріжджевих локусів HM типів спарювання. На відміну від SAGA, комплекс NuA3 не рекрутується дріжджевими промоторами за допомогою кислих активаторів.
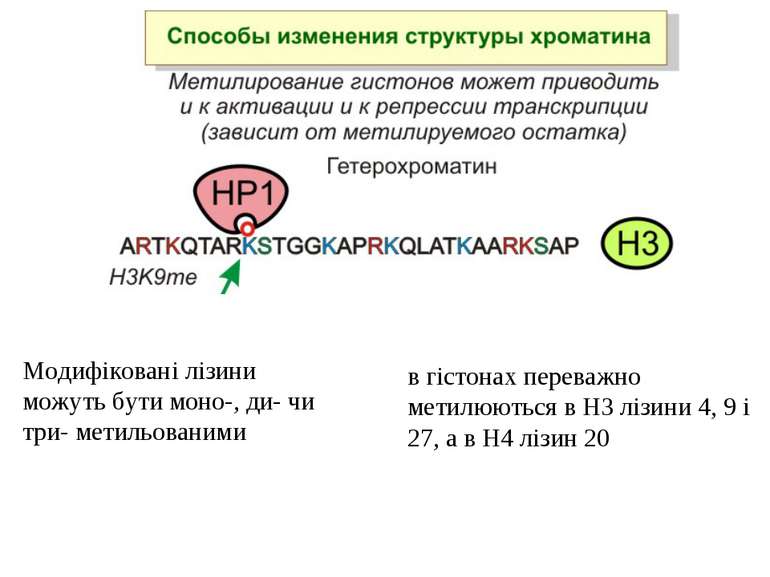
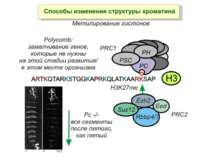
в гістонах переважно метилюються в H3 лізини 4, 9 і 27, а в H4 лізин 20 Модифіковані лізини можуть бути моно-, ди- чи три- метильованими
Фосфорилювання гістонів Тісно спряжене з ацетилюванням Н.: Ліз-9 Сер-10 Може відбувати у відповідь на пошкодження ДНК (ASQE-мотив Н2А)
NB !!! Метилювання і ацетилирювання Lys9 взаємовиключаючі. Метилювання Lys9 інгібує фосфорилювання Ser10, а не ацетилювання Lys14. Метилювання Lys9 корелює з мовчанням генів, тоді як фосфорилювання Ser10 і ацетилювання Lys9 корелює з транскрипційною активністю
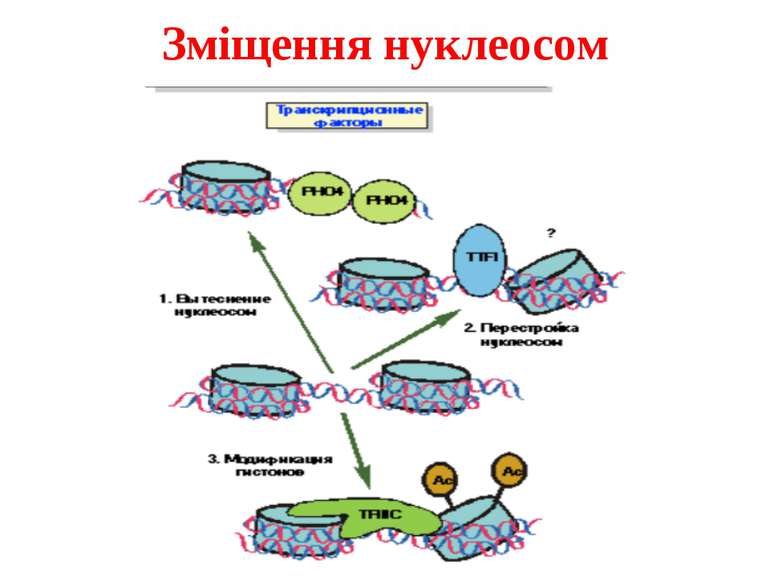
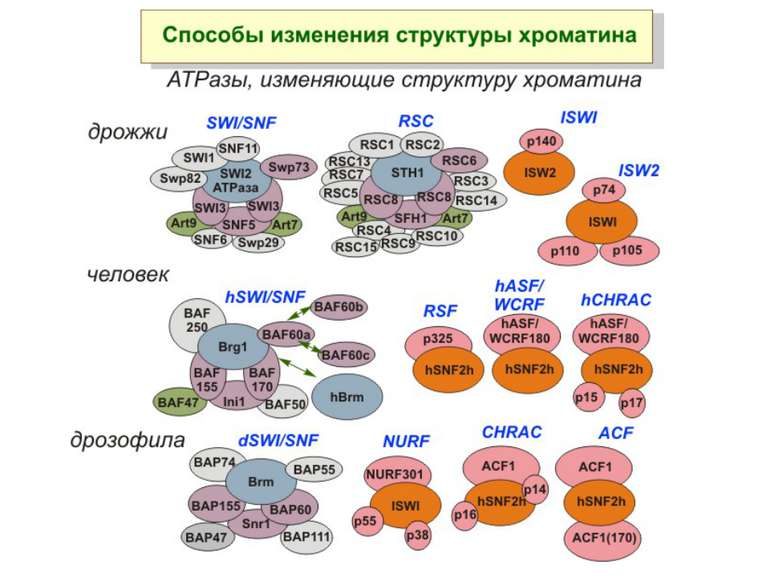
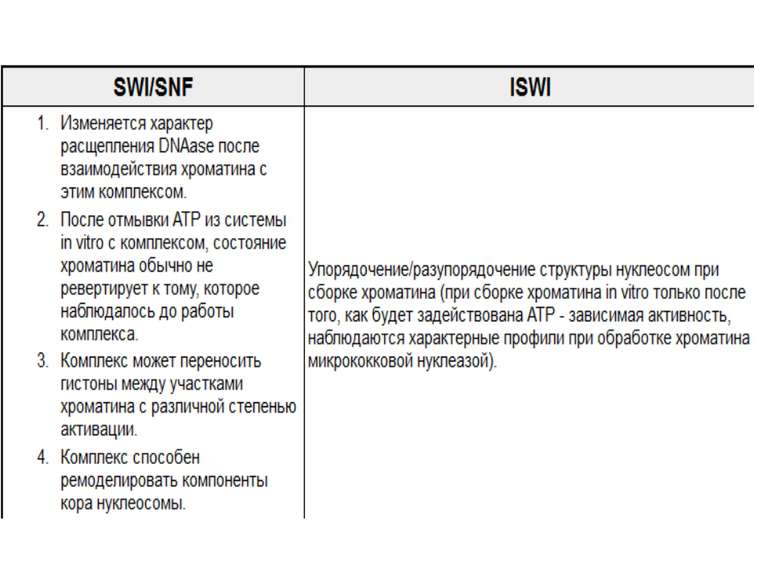
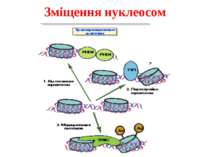
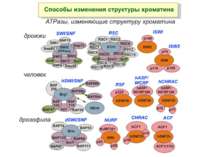
Комплекси, що містять гомологи SWI2 і ISWI відрізняються !!! SWI2 может використовувати як субстрат ненуклеосомну ДНК, і може виконувати складні перебудови ДНК-гістонового комплексу. Субстратом ISWI жуть бути лише нуклеосоми, а основною функцією – зсув нуклеосом по ДНК. Причому для ISWI необхідним є N-кінець гістону H4.
“Гістоновий код” різні паттерни модифікації гістонів (результат ацетилювання, фосфорилювання, метилювання і ін. пост-трансляційних модифікацій) діють послідовно або в комбінації для формування «гістонового коду», який зчитується за допомогою інших білків, щоб викликати певні низхідні транскрипційні події.
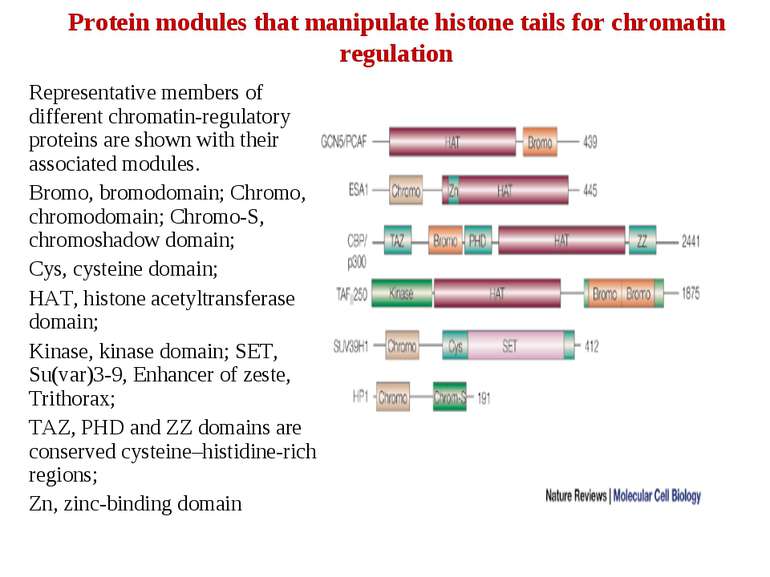
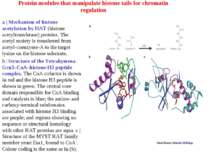
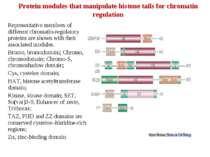
Protein modules that manipulate histone tails for chromatin regulation Representative members of different chromatin-regulatory proteins are shown with their associated modules. Bromo, bromodomain; Chromo, chromodomain; Chromo-S, chromoshadow domain; Cys, cysteine domain; HAT, histone acetyltransferase domain; Kinase, kinase domain; SET, Su(var)3-9, Enhancer of zeste, Trithorax; TAZ, PHD and ZZ domains are conserved cysteine–histidine-rich regions; Zn, zinc-binding domain
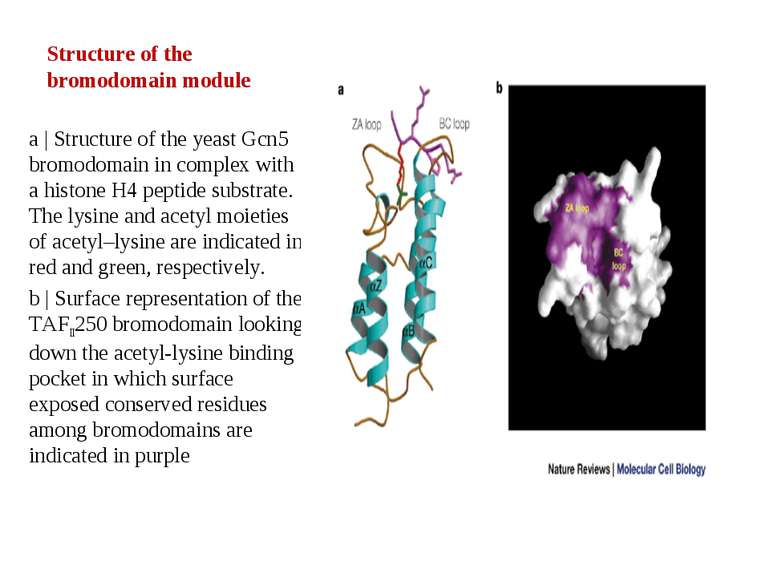
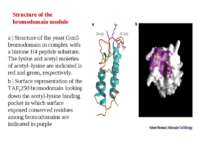
Structure of the bromodomain module a | Structure of the yeast Gcn5 bromodomain in complex with a histone H4 peptide substrate. The lysine and acetyl moieties of acetyl–lysine are indicated in red and green, respectively. b | Surface representation of the TAFII250 bromodomain looking down the acetyl-lysine binding pocket in which surface exposed conserved residues among bromodomains are indicated in purple
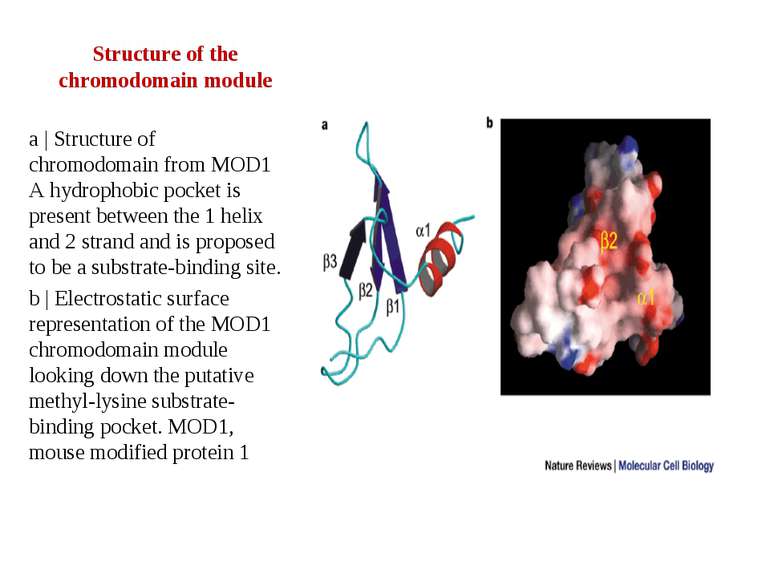
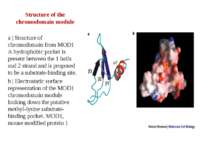
Structure of the chromodomain module a | Structure of chromodomain from MOD1 A hydrophobic pocket is present between the 1 helix and 2 strand and is proposed to be a substrate-binding site. b | Electrostatic surface representation of the MOD1 chromodomain module looking down the putative methyl-lysine substrate-binding pocket. MOD1, mouse modified protein 1
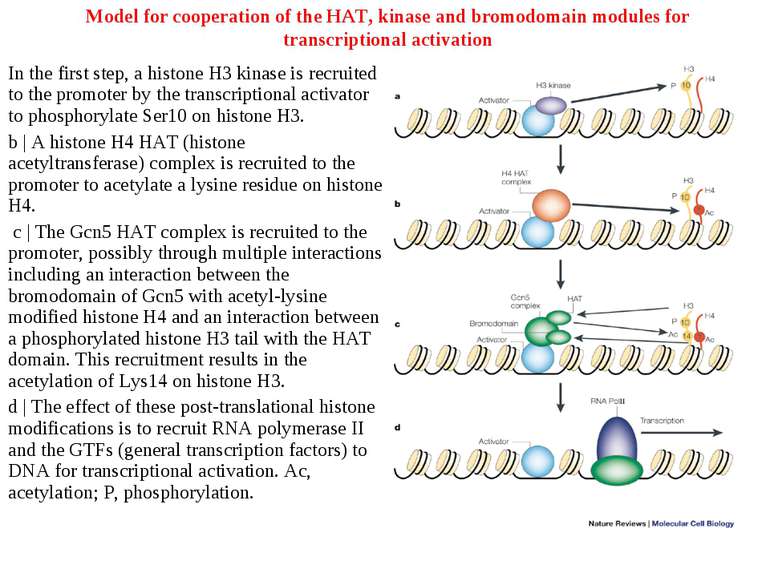
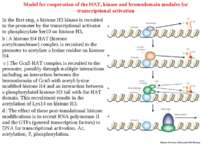
Model for cooperation of the HAT, kinase and bromodomain modules for transcriptional activation In the first step, a histone H3 kinase is recruited to the promoter by the transcriptional activator to phosphorylate Ser10 on histone H3. b | A histone H4 HAT (histone acetyltransferase) complex is recruited to the promoter to acetylate a lysine residue on histone H4. c | The Gcn5 HAT complex is recruited to the promoter, possibly through multiple interactions including an interaction between the bromodomain of Gcn5 with acetyl-lysine modified histone H4 and an interaction between a phosphorylated histone H3 tail with the HAT domain. This recruitment results in the acetylation of Lys14 on histone H3. d | The effect of these post-translational histone modifications is to recruit RNA polymerase II and the GTFs (general transcription factors) to DNA for transcriptional activation. Ac, acetylation; P, phosphorylation.
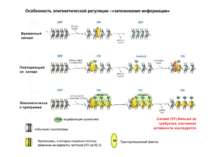
Після того, як відбулось ремоделювання структури хроматину HAC, SWI/SNF, ISWI комплексами, наступає час роботи РНК - полімерази II
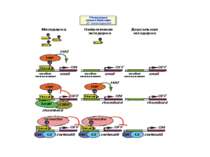
Моделі: На первом этапе с зарепрессированным хроматином связываются активаторы, которые способствуют работе над всей последовательностью гена ремоделирующих комплексов, которые, в свою очередь, делают ДНК доступной для работы РНК - полимеразы II. С зарепрессированным хроматином связываются активаторы. После этого вдоль последовательности гена проходит "первичная" РНК - полимераза, которая тащит на себе всё необходимое для ремоделирования хроматина. При этом полимераза транскрибирует ген. Имеются экспериментальные данные, указывающие на то, что первичный транскрипт отличается от последующих транскриптов с данного гена в данном активном состоянии. Вслед за "первичной" полимеразой начинает работать РНК - полимераза II, которая движется уже по ремоделированному хроматину вдоль последовательности гена.
Приклади с дрожжевым промотором HO сначала связывается SWI/SNF, затем SAGA. После этих хроматин-модифицирующих комплексов связывается активатор, привлекающий полимеразу за SRB/медиаторный комплекс (сидящий на CTD домене РНК полимеразы II). Совсем по другому происходит вызванная вирусом активация промотора интерферона. Сначала действует гистон-ацетилтрансфераза GCN5, затем гистон-ацетилтрансфераза CBP и происходит посадка РНК-полимеразы II. CBP привлекает SWI/SNF, который отодвигает нуклеосому от TATA последовательности и, таким образом, освобождает место для посадки TFIID. a1-антитрипсиновый промотор. В неактивном состоянии с ним связан активатор HNF-1a, TFIIB и TBP. При активации сначала собирается полный TFIID и присоединяется пролимераза, затем – гистонацетилтрансферазы CBP и PCAF. После гиперацетилирования гистонов в районе промотора, присоединяется HNF4 и SWI/SNF. После этого происходит инициация.
Системна регуляція еукаріоти Конформаційні перебудови хроматину Процесінг іРНК Утворення інформосом (аденілювання іРНК) Суперспіралізація ДНК – топоізомерази – зміна специфічності розпізнавання РНК П та сили зв’язування білків репресорів прокаріоти Зміна компонентного складу РНК-полімерази – синтез специфічних сигма-факторів бактеріофагами Дія гуанозинполіфосфатів Зміна кількості активних РНК-полімераз – негативна регуляція власного біосинтезу та перехід у латентний стан Транспозони
Схожі презентації
Категорії