Презентація на тему:
Якщо спадковість неоптимальна: редагування транскриптому, геному, мозку
Завантажити презентацію
Якщо спадковість неоптимальна: редагування транскриптому, геному, мозку
Завантажити презентаціюПрезентація по слайдам:
Якщо спадковість неоптимальна: редагування транскриптому, геному, мозку Болдирєв Олексій Інститут фізіології ім. О. О. Богомольця Міжнародний центр молекулярної фізіології НАН України evolution@science.ua my.science.ua
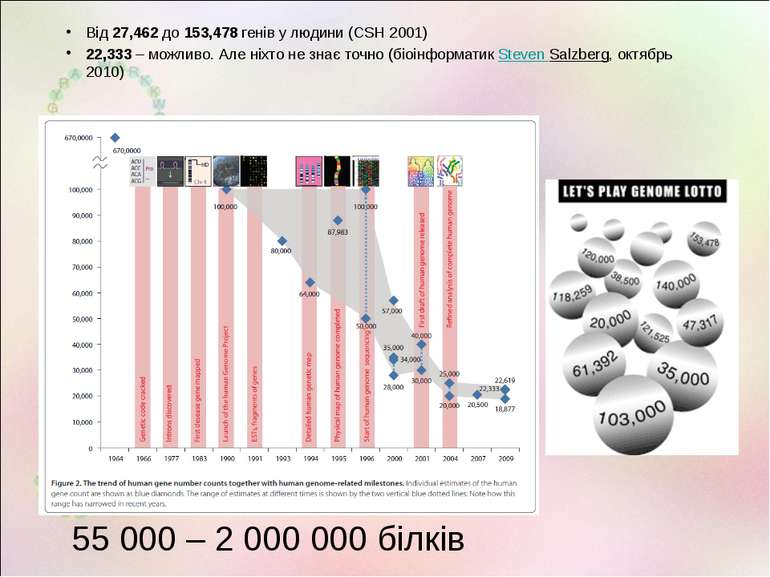
Від 27,462 до 153,478 генів у людини (CSH 2001) 22,333 – можливо. Але ніхто не знає точно (біоінформатик Steven Salzberg, октябрь 2010) 55 000 – 2 000 000 білків
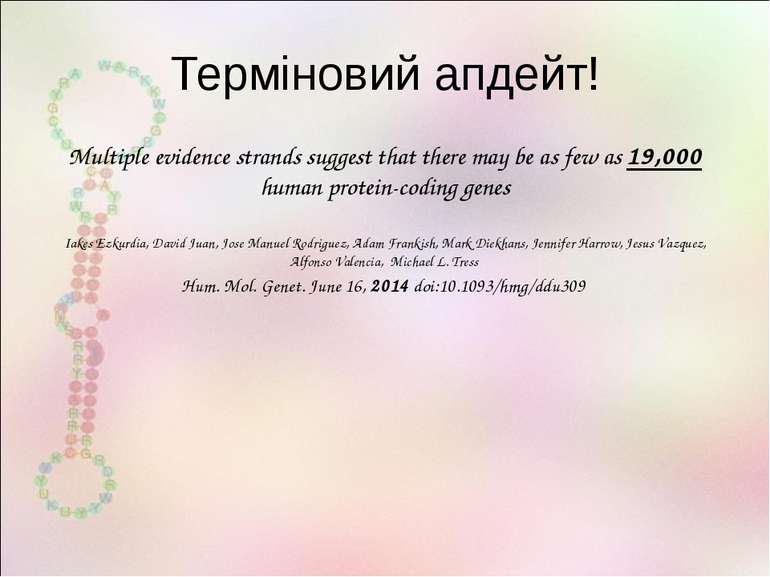
Терміновий апдейт! Multiple evidence strands suggest that there may be as few as 19,000 human protein-coding genes Iakes Ezkurdia, David Juan, Jose Manuel Rodriguez, Adam Frankish, Mark Diekhans, Jennifer Harrow, Jesus Vazquez, Alfonso Valencia, Michael L. Tress Hum. Mol. Genet. June 16, 2014 doi:10.1093/hmg/ddu309
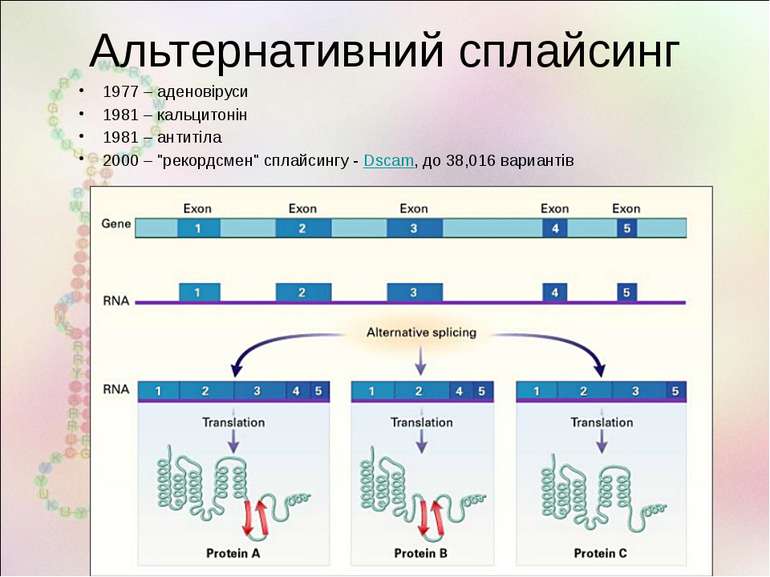
Альтернативний сплайсинг 1977 – аденовіруси 1981 – кальцитонін 1981 – антитіла 2000 – "рекордсмен" сплайсингу - Dscam, до 38,016 вариантів
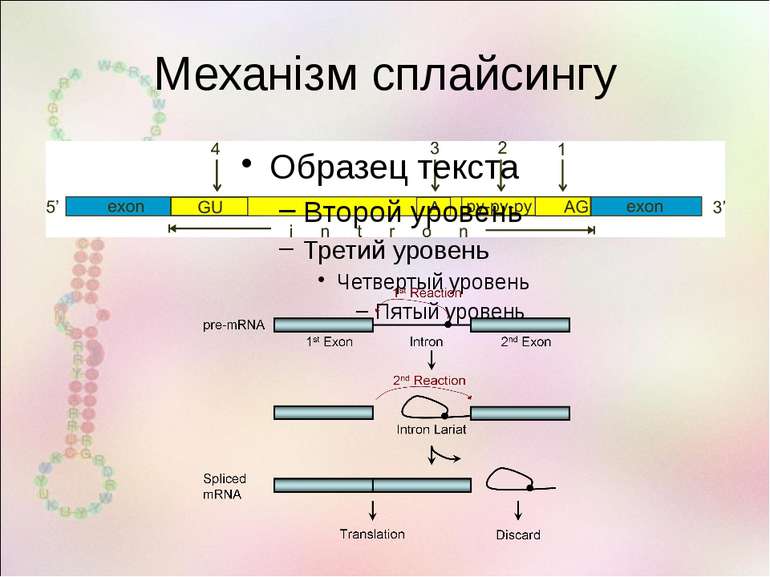
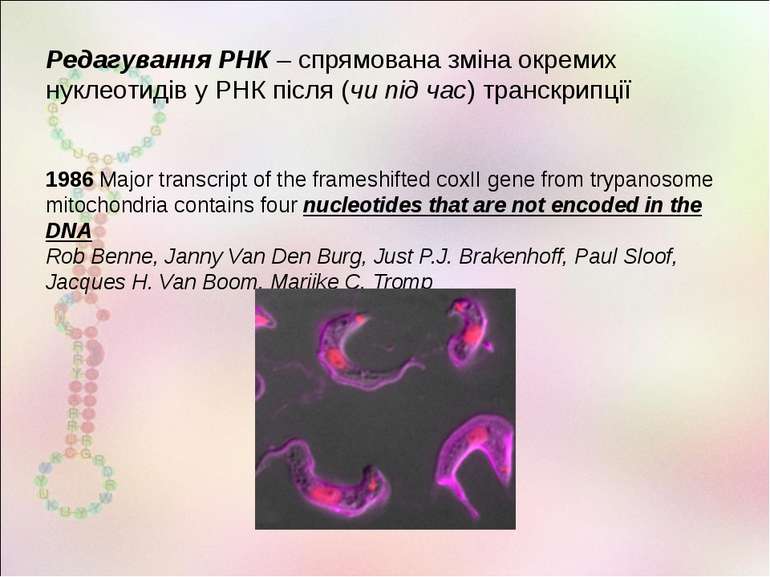
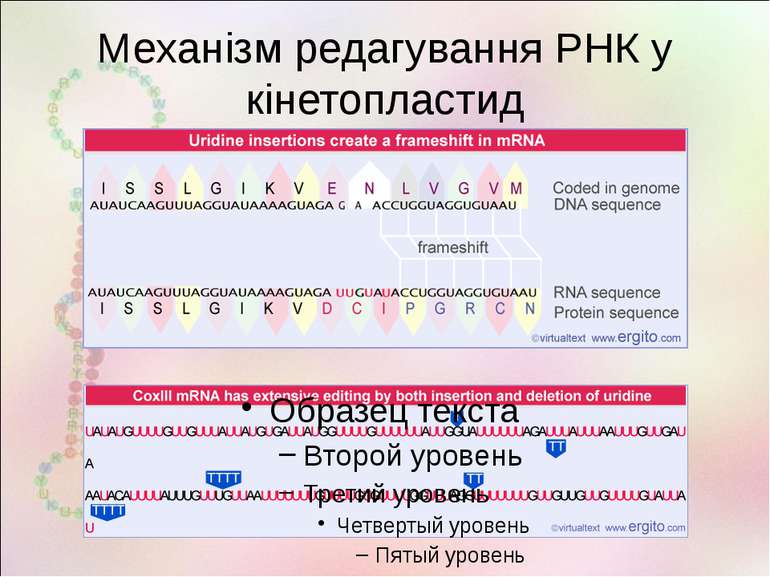
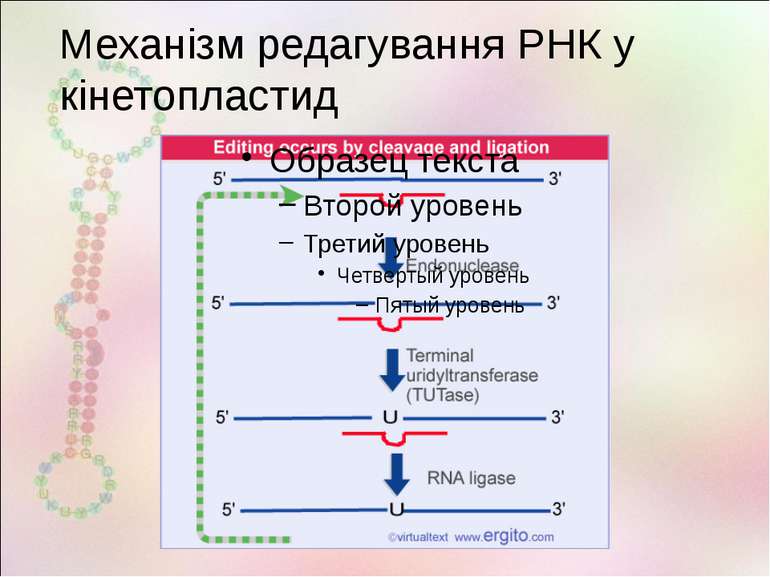
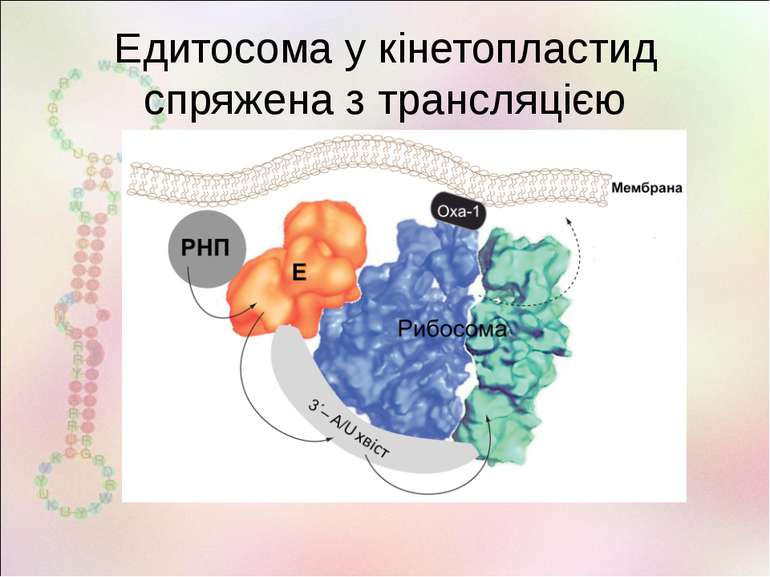
Редагування РНК – спрямована зміна окремих нуклеотидів у РНК після (чи під час) транскрипції 1986 Major transcript of the frameshifted coxII gene from trypanosome mitochondria contains four nucleotides that are not encoded in the DNA Rob Benne, Janny Van Den Burg, Just P.J. Brakenhoff, Paul Sloof, Jacques H. Van Boom, Marijke C. Tromp http://bioweb.wku.edu/courses/biol495g/CatalyticRNA27/RNAEditing5.html
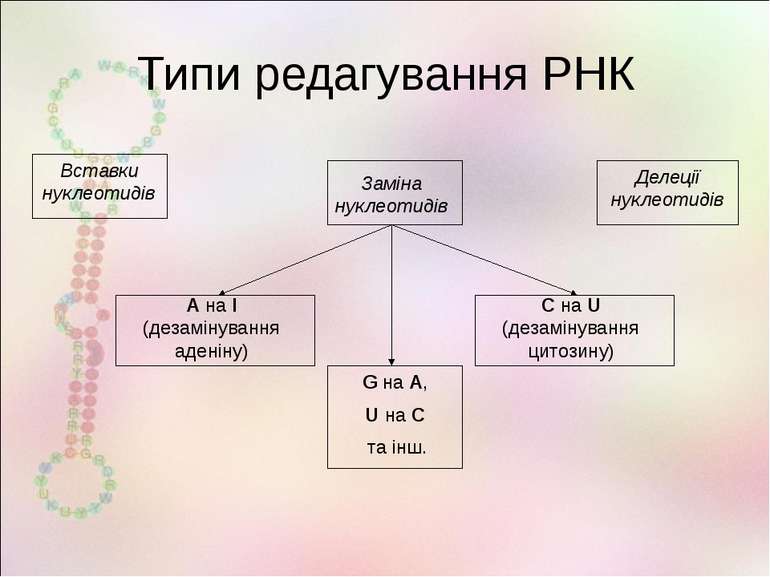
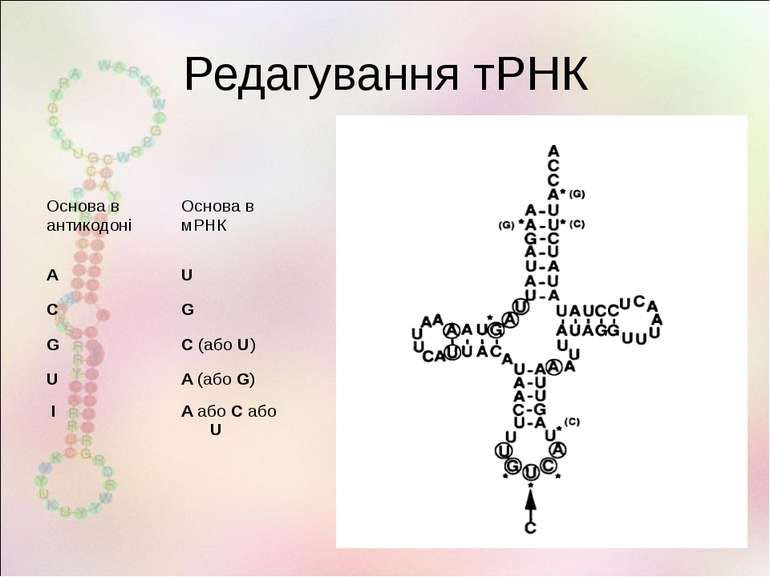
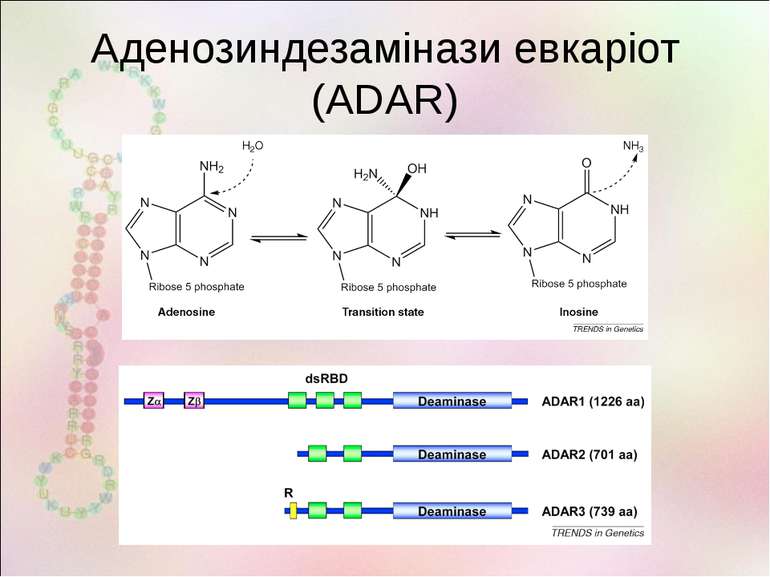
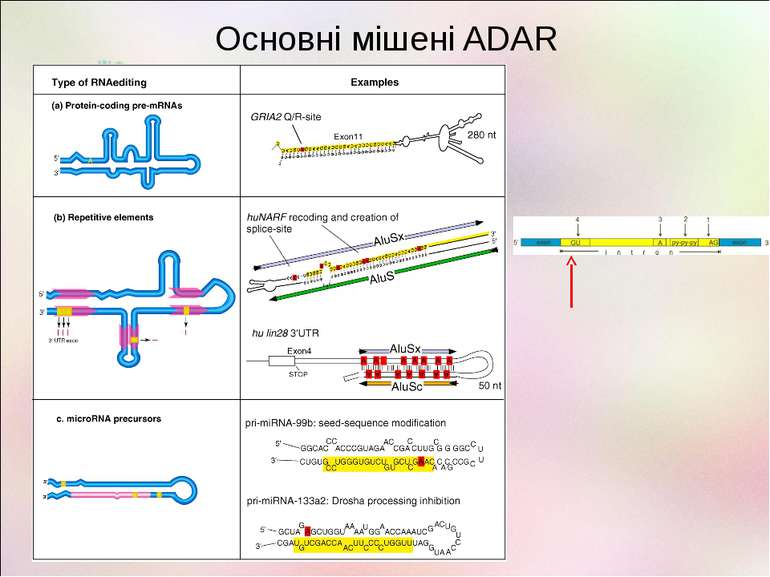
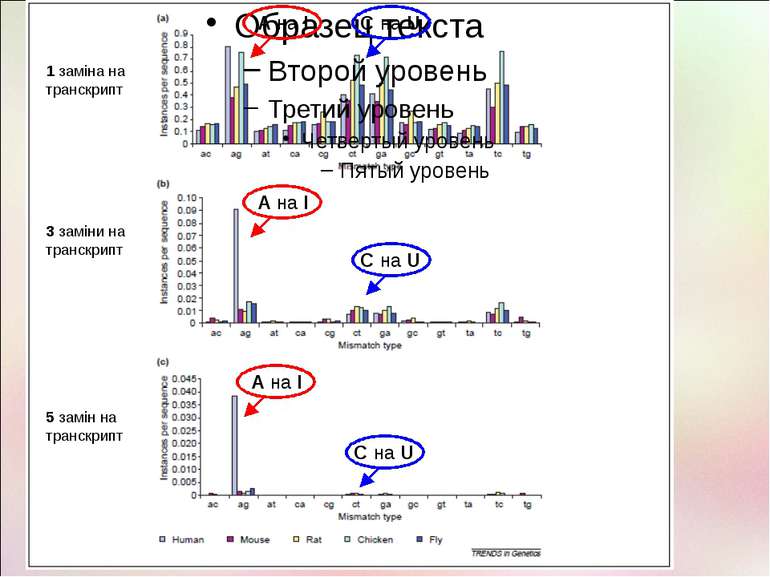
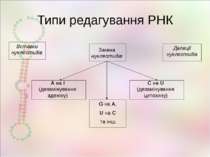
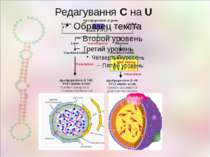
Типи редагування РНК А на І (дезамінування аденіну) С на U (дезамінування цитозину) G на A, U на C та інш. Вставки нуклеотидів Делеції нуклеотидів Заміна нуклеотидів
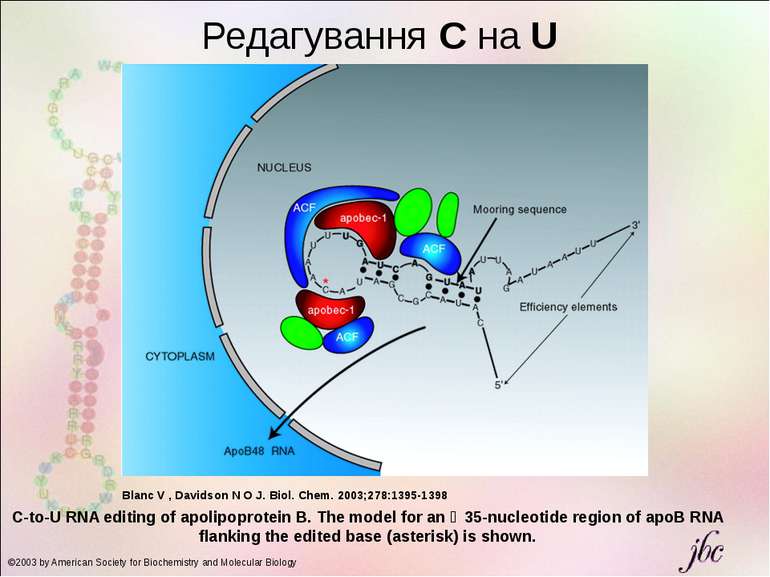
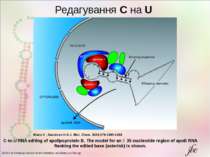
C-to-U RNA editing of apolipoprotein B. The model for an ∼35-nucleotide region of apoB RNA flanking the edited base (asterisk) is shown. Blanc V , Davidson N O J. Biol. Chem. 2003;278:1395-1398 ©2003 by American Society for Biochemistry and Molecular Biology Редагування С на U C-to-U RNA editing of apolipoprotein B.The model for an ∼35-nucleotide region of apoB RNA flanking the edited base (asterisk) is shown. A schematic representation illustrates apobec-1 (red) and ACF (blue) binding to RNA both 5′ and 3′ of the edited base and depicts the presence of additional proteins that may modulate assembly of the holoenzyme (green). Note that the stoichiometry of apobec-1 and ACF molecules with respect to the active enzyme is unknown. The model emphasizes the role of both cis-acting elements within the vicinity of the edited base (mooring sequence isbolded) and the requirement for an optimal structure, conferred by both 5′ and 3′ efficiency elements.
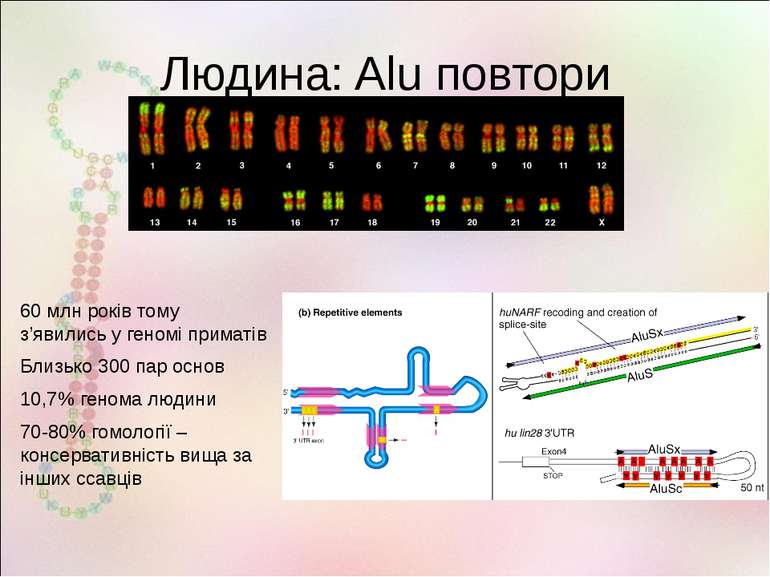
Людина: Alu повтори 60 млн років тому з’явились у геномі приматів Близько 300 пар основ 10,7% генома людини 70-80% гомології –консервативність вища за інших ссавців
У Редактора РНК нема нічого святого! И все це впорядковано працює в організмі, а порушення всіх цих процесів призводить до ВЕЛИКИХ неприємностей!!!! Код ДНК не гарантує амінокислотної послідовністі білка: з’являються стоп-кодони, заміняються амінокислоти Сплайсинг відбувається «як прийдеться»: нові сайти виникають, старі зникають тРНК, яка створює «не ті» білки «Сміттєва ДНК» активно використовується для роботи еволюційно просунутих видів (людина й примати) і органів (тимус, мозок)
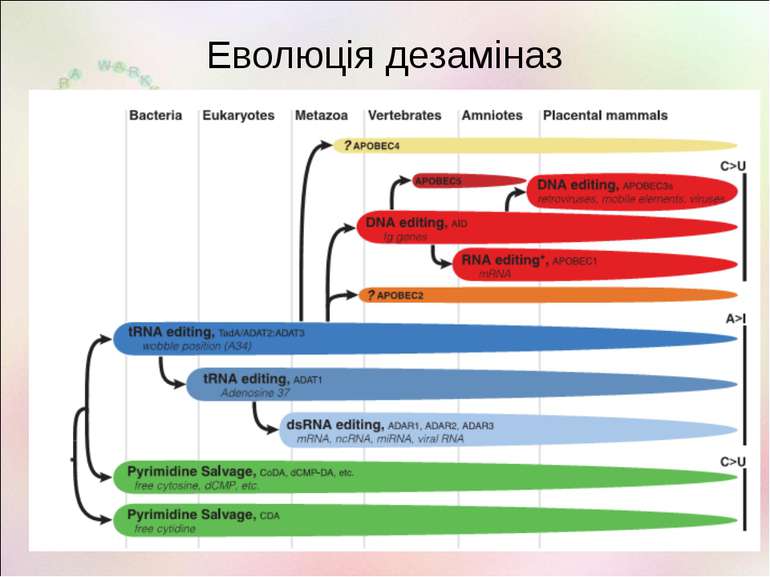
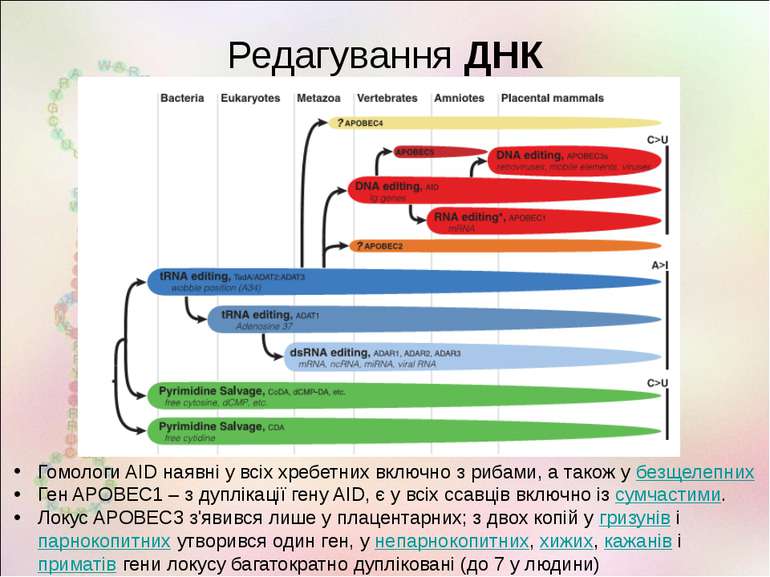
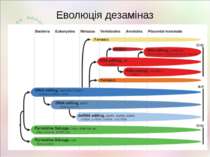
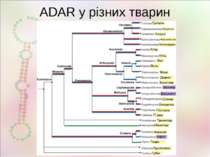
Редагування ДНК Гомологи AID наявні у всіх хребетних включно з рибами, а також у безщелепних Ген APOBEC1 – з дуплікації гену AID, є у всіх ссавців включно із сумчастими. Локус APOBEC3 з'явився лише у плацентарних; з двох копій у гризунів і парнокопитних утворився один ген, у непарнокопитних, хижих, кажанів і приматів гени локусу багатократно дупліковані (до 7 у людини) http://users.rcn.com/jkimball.ma.ultranet/BiologyPages/R/RNA_Editing.html
Гени родини APOBEC у людини AID 12p13 5 B-лімфоцити,сім'яники Цитоплазматичнийбілок,працюєуядрі ДезамінуванняДНК,гениімуноглобулінів APOBEC1 12p13.1 5 Тонкий кишечник (уіншихссавців — кишечник) Цитоплазматичнийбілок,працюєуядрі ДезамінуваннямРНКаполіпопротеїнуB,дезамінуванняДНК APOBEC2 6p21 3 Скелетнім'язи,серце Цитоплазматичнийіядернийбілок Береучасть вембріогенезім'язів,мішеніневідомі APOBEC3A 22q13.1 5 Кератиноцити,клітиникрові Цитоплазматичнийіядернийбілок ДезамінуванняДНКаденоасоційованихвірусів,ретротранспозонів. APOBEC3B 22q13.1 8 Кишечник,матка,молочназалоза,кератиноцититаінші Переважноядернийбілок ДезамінуванняДНКретровірусів,вірусугепатитуB APOBEC3C 22q13.1 4 Убагатьохтканинах Цитоплазматичнийіядернийбілок ДезамінуванняДНКретровірусів,вірусугепатитуB,ретротранспозонів APOBEC3DE 22q13.1 7 Щитоподібназалоза,селезінка,лейкоцити Невідомо ДезамінуванняДНКретровірусів APOBEC3F 22q13.1 8 Убагатьохтканинах Цитоплазматичнийбілок ДезамінуванняДНКретровірусів,вірусугепатитуB,ретротранспозонів APOBEC3G 22q13.1 8 Убагатьохтканинах,Т-лімфоцитах Цитоплазматичнийбілок ДезамінуванняДНКретровірусів,вірусугепатитуB,ретротранспозонів APOBEC3H 22q13.1 5 Лейкоцити,тимус,щитоподібназалоза, плацента Невідомо ДезамінуванняДНКретровірусів APOBEC4 1q25.3 2 Сім'яники Невідомо Невідомі
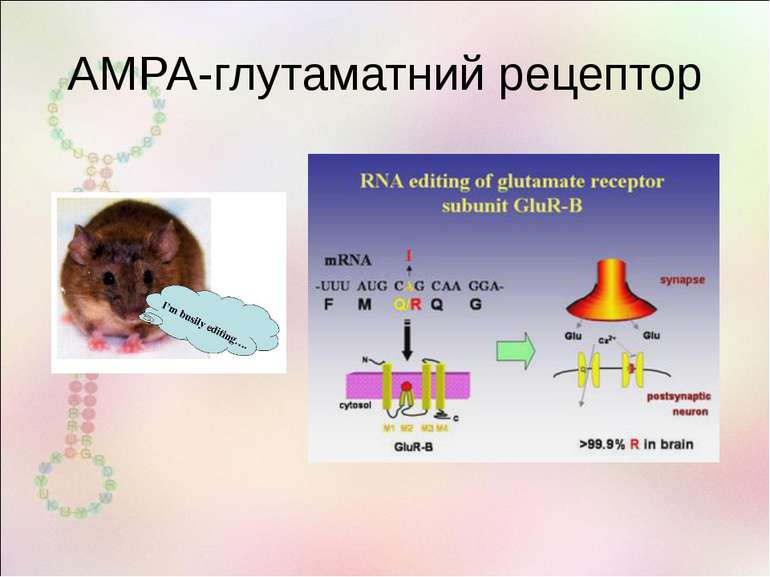
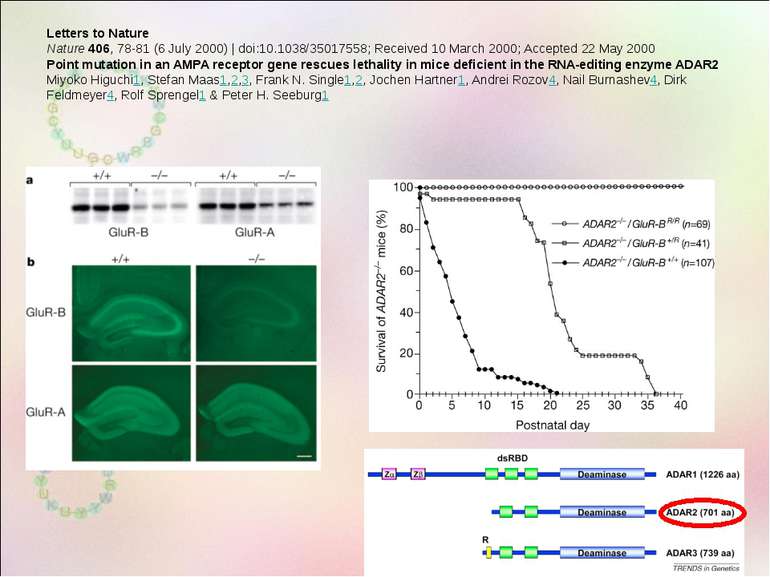
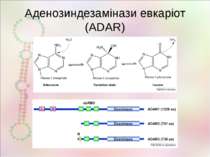
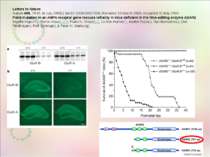
Letters to Nature Nature 406, 78-81 (6 July 2000) | doi:10.1038/35017558; Received 10 March 2000; Accepted 22 May 2000 Point mutation in an AMPA receptor gene rescues lethality in mice deficient in the RNA-editing enzyme ADAR2 Miyoko Higuchi1, Stefan Maas1,2,3, Frank N. Single1,2, Jochen Hartner1, Andrei Rozov4, Nail Burnashev4, Dirk Feldmeyer4, Rolf Sprengel1 & Peter H. Seeburg1
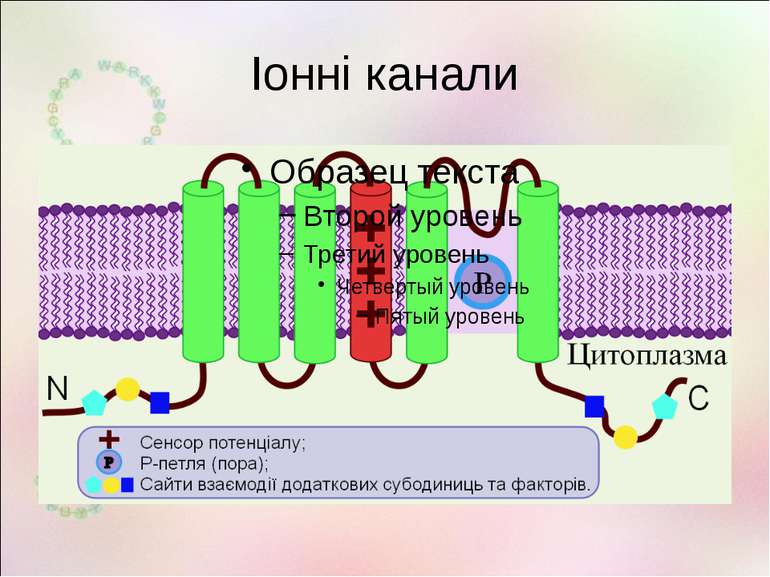
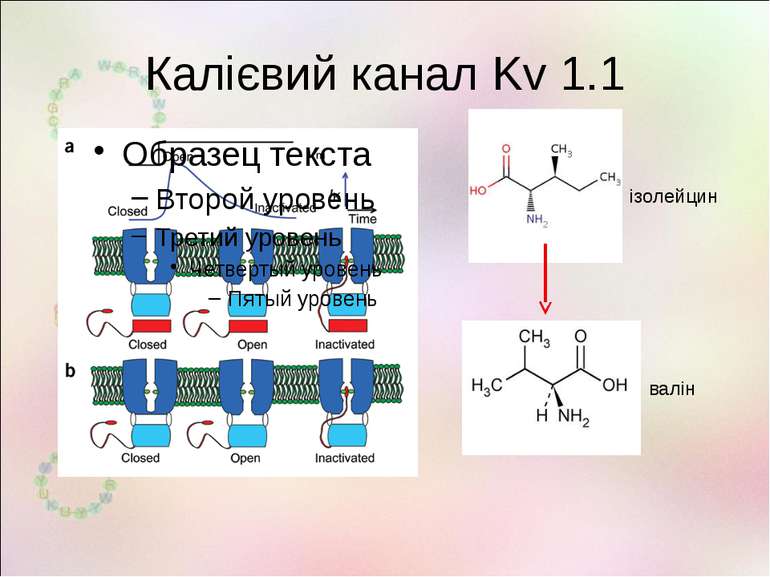
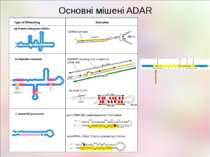
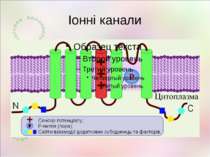
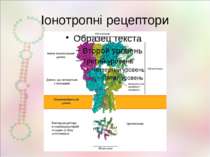
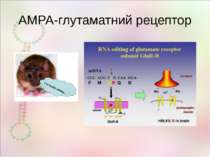
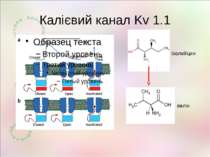
Редагування РНК у нервовій системі Описанівипадкиредагуваннявекзонах,якізмінюютьфункціюбілка GluR2 (Gria2,AMPA2) Q/R R/G ГлутаматнийAMPA-рецептор зіоннимканалом мозок Непроникність каналу для кальцію GluR3 R/G ГлутаматнийAMPA-рецептор зіоннимканалом мозок Прискореневідновленняпіслядесенсетизації GluR4 R/G ГлутаматнийAMPA-рецептор зіоннимканалом мозок Прискорене відновлення після десенсетизації GluR5 (Grik1) Q/R Глутаматнийкаїнатнийрецептор зіоннимканалом мозок Непроникність каналу для кальцію GluR6 (Grik2) Q/R I/V Y/C Глутаматнийкаїнатнийрецептор зіоннимканалом мозок Зміна проникності для іонів KCNA1 I/V Потенціал-залежнийкалієвийканал мозок Уповільнена інактивація каналу Gabra3 K/E РецепторГАМКзіоннимканалом мозок, кишечник ЗмінаспорідненостідоГАМК 5HT2cR I/V,I/M,N/S,N/DN/G I/V Серотоніновийрецептор мозок Змінене зв'язування із серотоніном, змінений транспорт рецептора IGFBP7 K/R R/G Інсуліновийрецептор,регуляціяросту йміграціїклітингліоми мозок,глія змінюєзв'язуваннязгепарином
Редагування РНК у нервовій системі KCNMA1 S/G Кальційзалежнийкалієвийканалвеликоїпровідності ? KIF20B K/R Кінезиновийбілок, транспорт помікротрубках,цитокінез всюди ? GANAB Q/R Глюкозидаза,відрізаєN-зв'язанігліканивідбілків ? OS9 E/G Глікопротеїнсистемидеградації,пов'язаноїзретикулумом ? STRN4 (стріатин,зінедин) R/G Білокцитоскелету,кальмодулін-зв'язуючийбілок, передача сигналу в нейронах нейрони ? BIN1 (амфіфізинII) K/R Пухлиннийсупресор,кардіоміопатія мозок ? SS18L1 S/G Кальційзалежнийактиватортранскрипції,необхіднийдля нормального росту йгалуженнядендритіву нейронах кори нейроникори головногомозку ? CADPS E/G Кальційзалежнийактиваторсекреціїйекзоцитозущільнихвезикул ? ATXN7 (атаксин7) K/R Субодиницяацетил-трансферазигістонів мозок ? Elavl2 I/V N/D Нейрон-специфічнийРНК-зв'язуючийбілок,диференціаціянейронівісинаптичнапластичність нейрони ?
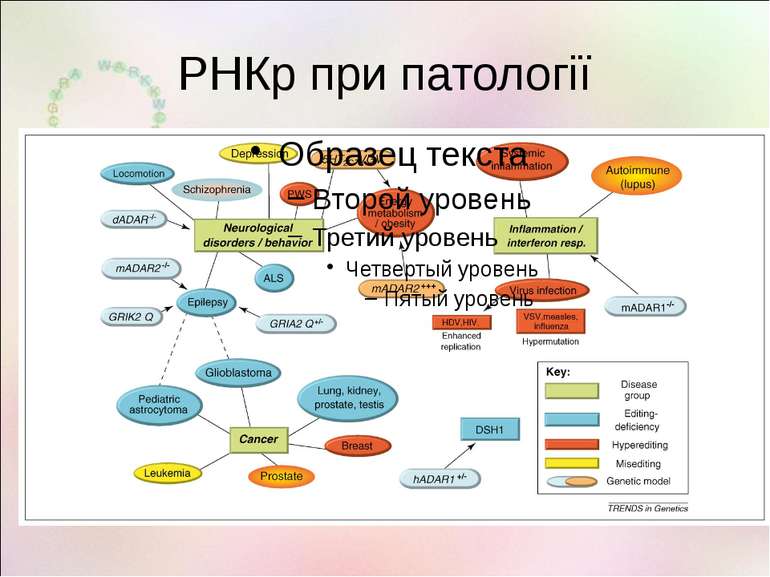
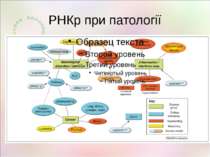
Кількість сайтів редагування в екзонах 115 сайтів ADAR 53 сайти ADAR 645 сайтів ADAR у нервовій системі – 60% білків!
Головний Редактор РНК: Модифікує функцію білка Знищує непотрібну РНК Бореться з вірусами Тканиноспецифічно змінює експресію білка Керує РНК-інтерференцією Впливає на епігенетичну адаптацію й еволюцію Регулює сплайсинг білка
Література Molecular diversity through RNA editing: a balancing act Sanaz Farajollahi and Stefan Maas Trends in Genetics Vol.26 No.5 (2010) RNA editing: a driving force for adaptive evolution? Willemijn M. Gommans, Sean P. Mullen, Stefan Maas Bioessays. 2009 October ; 31(10): 1137–1145. Is abundant A-to-I RNA editing primate-specific? Eli Eisenberg, Sergey Nemzer, Yaron Kinar, Rotem Sorek, Gideon Rechavi and Erez Y. Levanon TRENDS in Genetics Vol.21 No.2 (2005) БІЛЬШЕ ПОСИЛАНЬ: http://uk.wikipedia.org/wiki/Редагування_РНК
Схожі презентації
Категорії