Презентація на тему:
Translation_control_procariot 1 11 11
Завантажити презентацію
Translation_control_procariot 1 11 11
Завантажити презентаціюПрезентація по слайдам:
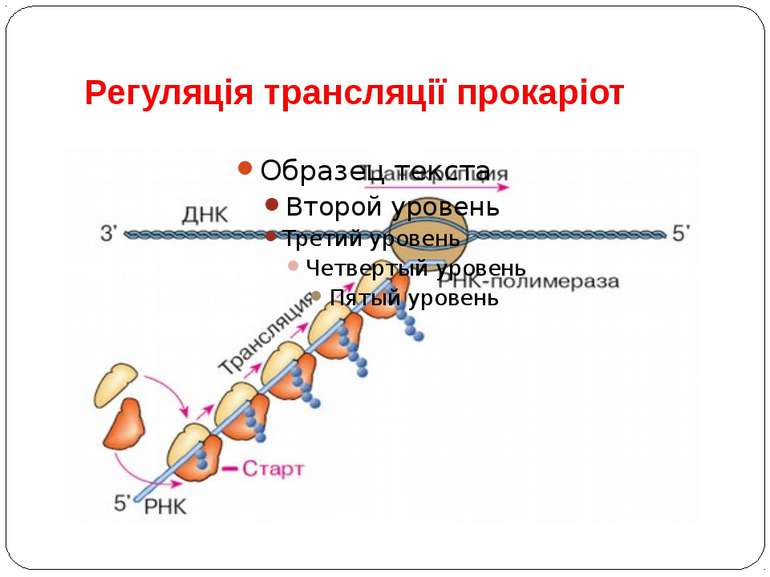
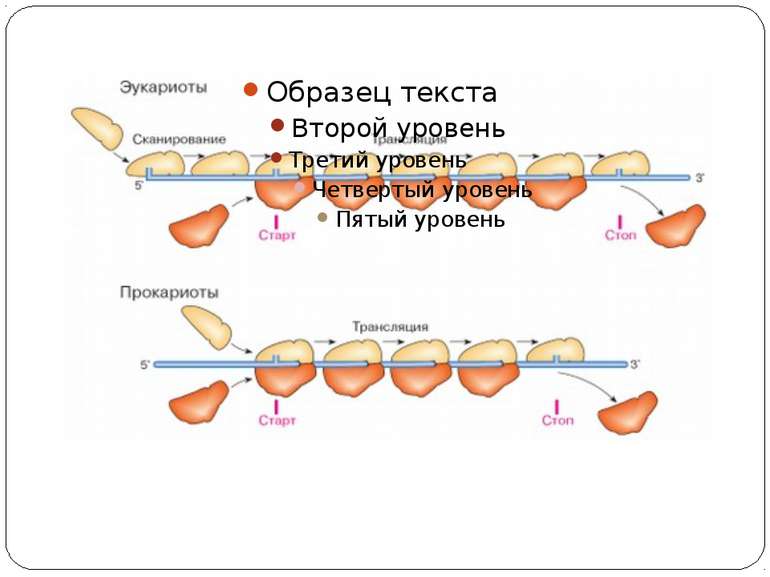
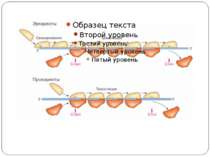
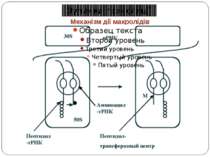
Рівні регуляції трансляції прокаріот На етапі біосинтезу і збирання компонентів апарату трансляції На етапі функціонування апарату трансляції (ініціація, елонгація, термінація)
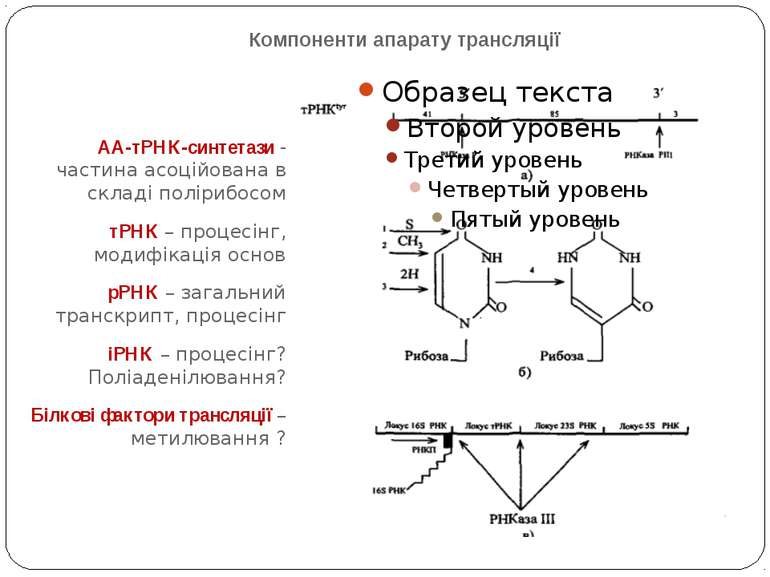
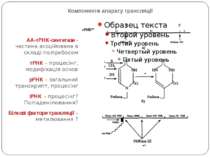
Компоненти апарату трансляції АА-тРНК-синтетази - частина асоційована в складі полірибосом тРНК – процесінг, модифікація основ рРНК – загальний транскрипт, процесінг іРНК – процесінг? Поліаденілювання? Білкові фактори трансляції – метилювання ?
Регуляція ініціації трансляції Контроль стабільності мРНК Позитивна регуляція на основі спорідненості мРНК до ініціюючої рибосоми й факторів ініціації (дискримінація мРНК). Негативна регуляція за допомогою білків-репресорів, які, зв'язуючись із мРНК, блокують ініціацію (трансляційна репресія) Трансляційне спряження
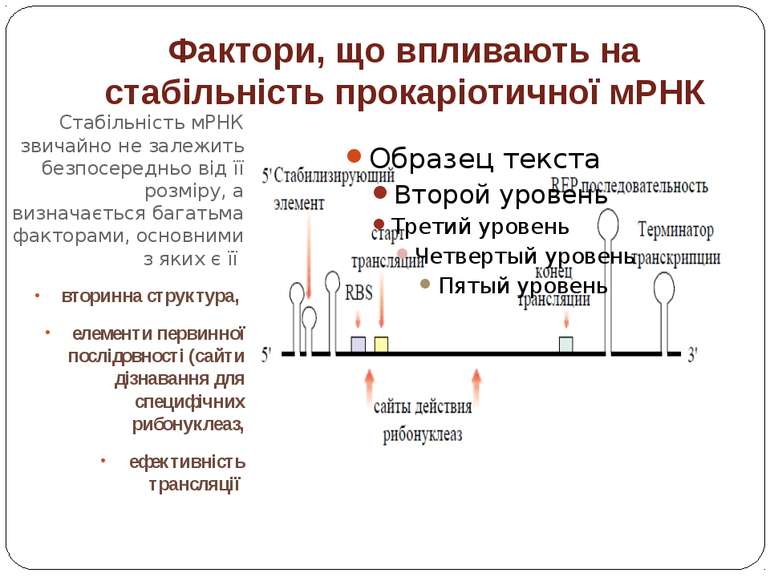
Фактори, що впливають на стабільність прокаріотичної мРНК Стабільність мРНК звичайно не залежить безпосередньо від її розміру, а визначається багатьма факторами, основними з яких є її вторинна структура, елементи первинної послідовності (сайти дізнавання для специфічних рибонуклеаз, ефективність трансляції
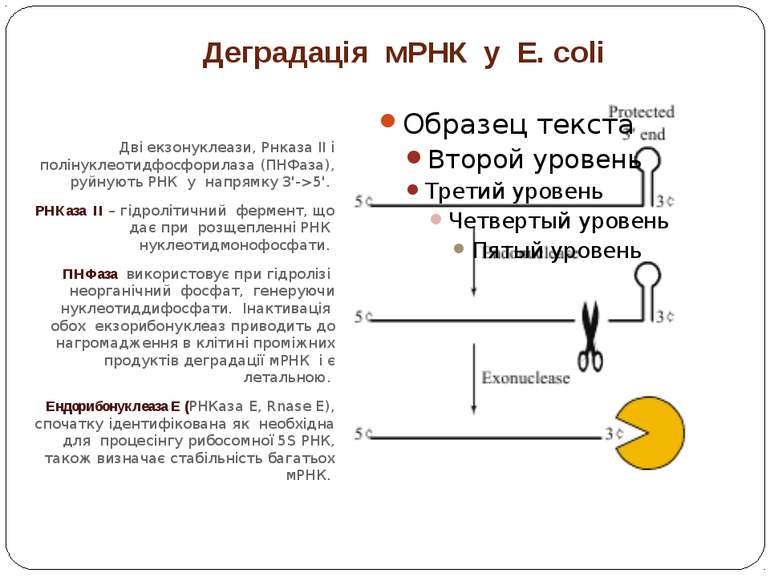
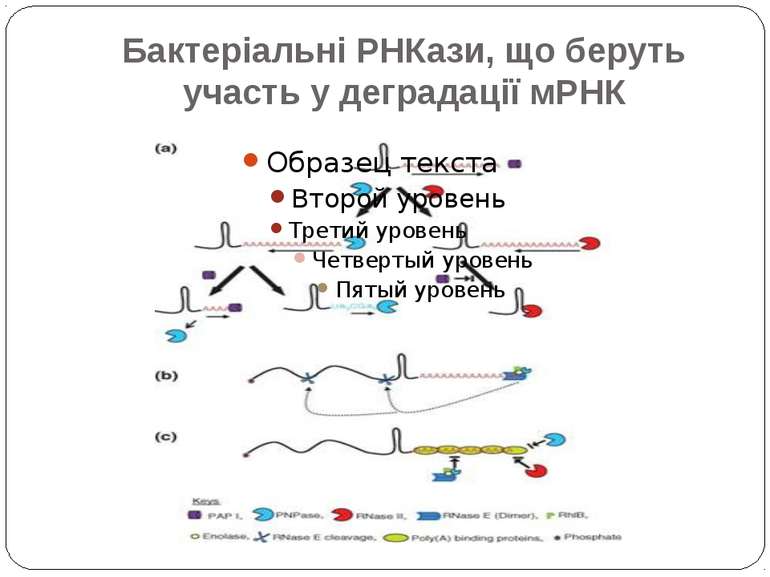
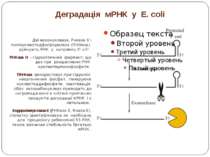
Деградація мРНК у E. coli Дві екзонуклеази, Рнказа II і полінуклеотидфосфорилаза (ПНФаза), руйнують РНК у напрямку 3'->5'. РНКаза II – гідролітичний фермент, що дає при розщепленні РНК нуклеотидмонофосфати. ПНФаза використовує при гідролізі неорганічний фосфат, генеруючи нуклеотиддифосфати. Інактивація обох екзорибонуклеаз приводить до нагромадження в клітині проміжних продуктів деградації мРНК і є летальною. Ендорибонуклеаза E (РНКаза E, Rnase E), спочатку ідентифікована як необхідна для процесінгу рибосомної 5S РНК, також визначає стабільність багатьох мРНК.
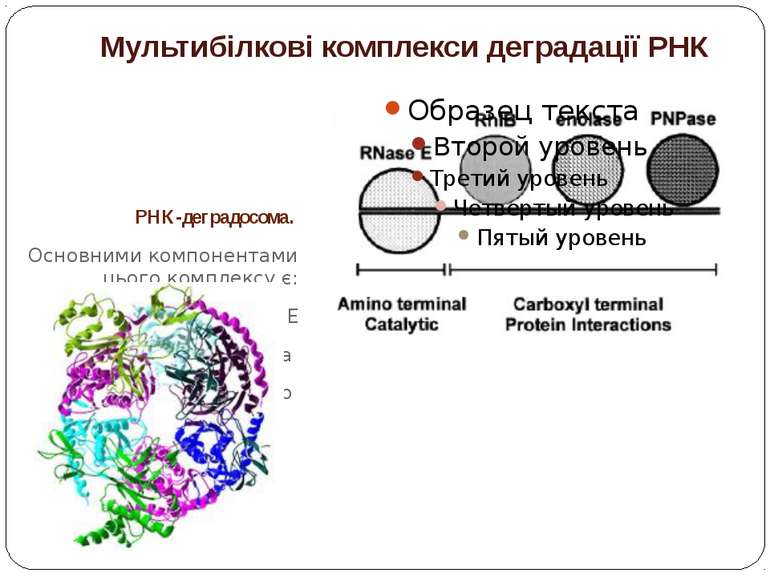
Мультибілкові комплекси деградації РНК РНК-деградосома. Основними компонентами цього комплексу є: РНКаза Е ПНФаза РНК-хеліказа Rhlb
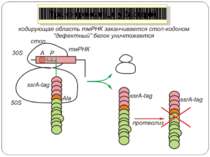
Поліаденілювання проявляє протилежний ефект на стабільність бактеріальних і еукаріотичних мРНК Додавання полі(А) сприяє деградації РНК, чиї кінці захищені від дії екзонуклеаз вторинною структурою. Такі екзорибонуклеази, як ПНФаза й РНКаза I, потребують одноланцюговий "хвіст", за який можна "схопити" 3' кінець мРНК і почати її деградацію. Поліаденілювання 3' кінця створює сайт зв'язування для екзорибонуклеаз
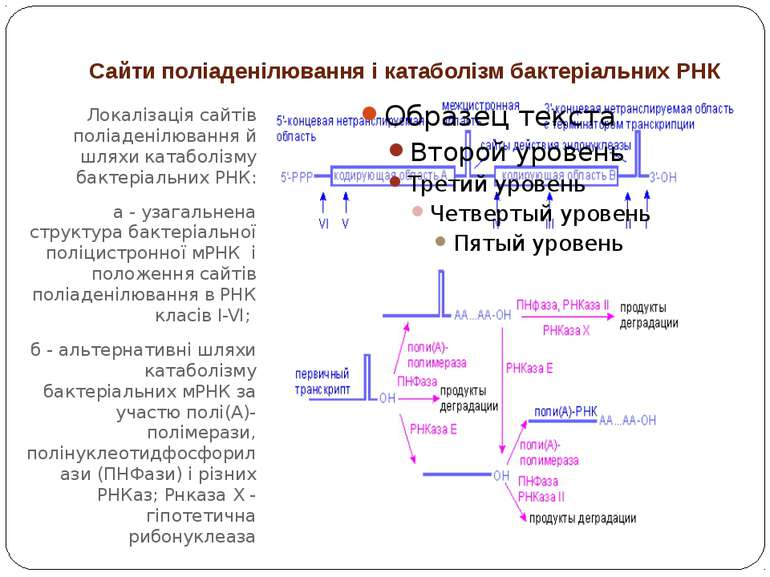
Сайти поліаденілювання і катаболізм бактеріальних РНК Локалізація сайтів поліаденілювання й шляхи катаболізму бактеріальних РНК: а - узагальнена структура бактеріальної поліцистронної мРНК і положення сайтів поліаденілювання в РНК класів I-VI; б - альтернативні шляхи катаболізму бактеріальних мРНК за участю полі(А)-полімерази, полінуклеотидфосфорилази (ПНФази) і різних РНКаз; Рнказа Х - гіпотетична рибонуклеаза
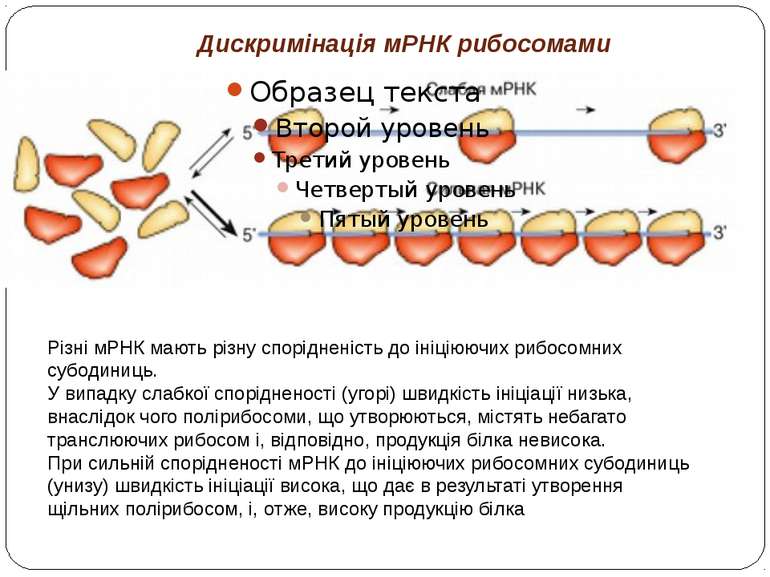
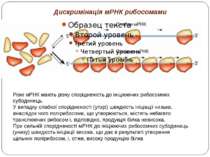
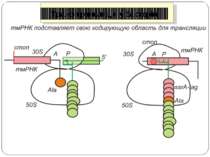
Дискримінація мРНК рибосомами Різні мРНК мають різну спорідненість до ініціюючих рибосомних субодиниць. У випадку слабкої спорідненості (угорі) швидкість ініціації низька, внаслідок чого полірибосоми, що утворюються, містять небагато транслюючих рибосом і, відповідно, продукція білка невисока. При сильній спорідненості мРНК до ініціюючих рибосомних субодиниць (унизу) швидкість ініціації висока, що дає в результаті утворення щільних полірибосом, і, отже, високу продукцію білка
Дискримінація мРНК рибосомами дискримінацію мРНК можна розглядати як механізм конститутивного контролю належного фіксованого співвідношення продуктів білкового синтезу Н.: мембранний комплекс протонної АТФази бактерій побудований із трьох типів субодиниць: a, b і c - у співвідношенні 1:2:10 (a1b2c10), і відповідно субодиниця c кодується дуже сильним цистроном мРНК, субодиниця a - слабким, а субодиниця b - цистроном проміжної сили
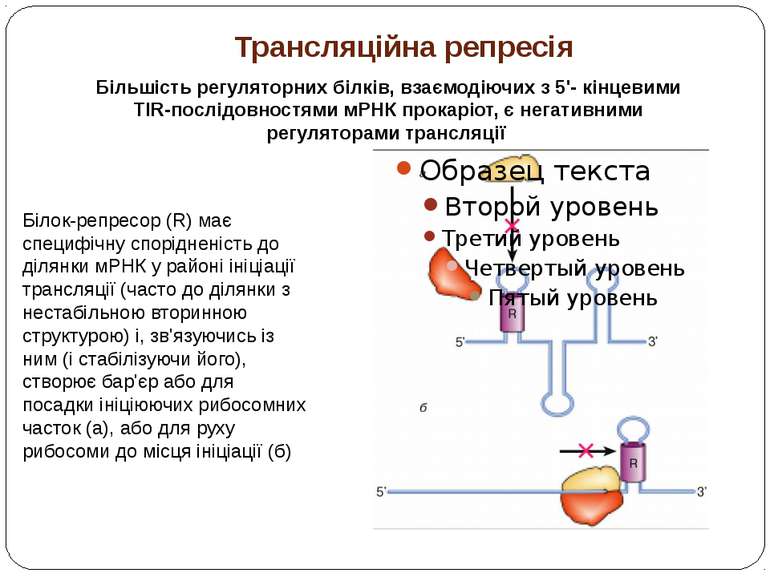
Трансляційна репресія Білок-репресор (R) має специфічну спорідненість до ділянки мРНК у районі ініціації трансляції (часто до ділянки з нестабільною вторинною структурою) і, зв'язуючись із ним (і стабілізуючи його), створює бар'єр або для посадки ініціюючих рибосомних часток (а), або для руху рибосоми до місця ініціації (б) Більшість регуляторних білків, взаємодіючих з 5'- кінцевими TIR-послідовностями мРНК прокаріот, є негативними регуляторами трансляції
Трансляційна репресія Рибосомні білки E. coli - запобігають взаємодії 30S субодиниць рибосом зі своїми мРНК рибосомний білок S15 - взаємодіючи з TIR-послідовністю своєї мРНК стабілізує преіснуючий псевдовузел. У результаті SD-область мРНК стає пасткою для 30S субодиниці рибосоми, яка взаємодіє з нею, але не може ініціювати синтез білка.
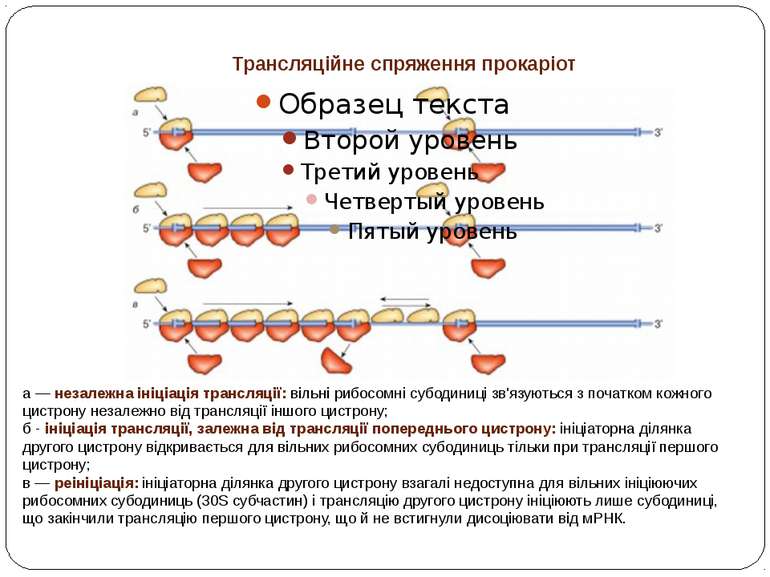
Трансляційне спряження прокаріот а — незалежна ініціація трансляції: вільні рибосомні субодиниці зв'язуються з початком кожного цистрону незалежно від трансляції іншого цистрону; б - ініціація трансляції, залежна від трансляції попереднього цистрону: ініціаторна ділянка другого цистрону відкривається для вільних рибосомних субодиниць тільки при трансляції першого цистрону; в — реініціація: ініціаторна ділянка другого цистрону взагалі недоступна для вільних ініціюючих рибосомних субодиниць (30S субчастин) і трансляцію другого цистрону ініціюють лише субодиниці, що закінчили трансляцію першого цистрону, що й не встигнули дисоціювати від мРНК.
Умови реініціації в сайті реініціації є адекватна SD-послідовність термінуючий і ініціюючий кодон розміщені достатньо близько один до іншого (менше еквіваленту довжини рибосоми) Часто на UUG –кодоні (відсутність в комплексі IF3
Антисмислові РНК є потужними природними модуляторами експресії генів у прокаріот Прокаріотичні антисмислові РНК довжиною 70-110 нп утворюють структури типу “стебло-петля", у яких стебло захищає ці РНК від деградації, а петля довжиною шість-вісім нп служить для початкової взаємодії із мРНК-мішенню. Після утворенні комплексів РНК-РНК спостерігали зміну стабільності мРНК, ефективності процесінгу Рнк-мішені, термінації транскрипції або ініціації їх трансляції
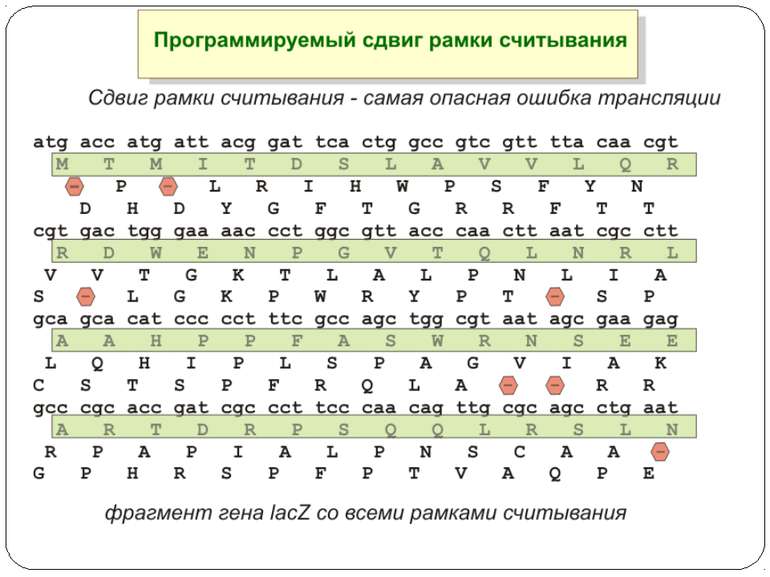
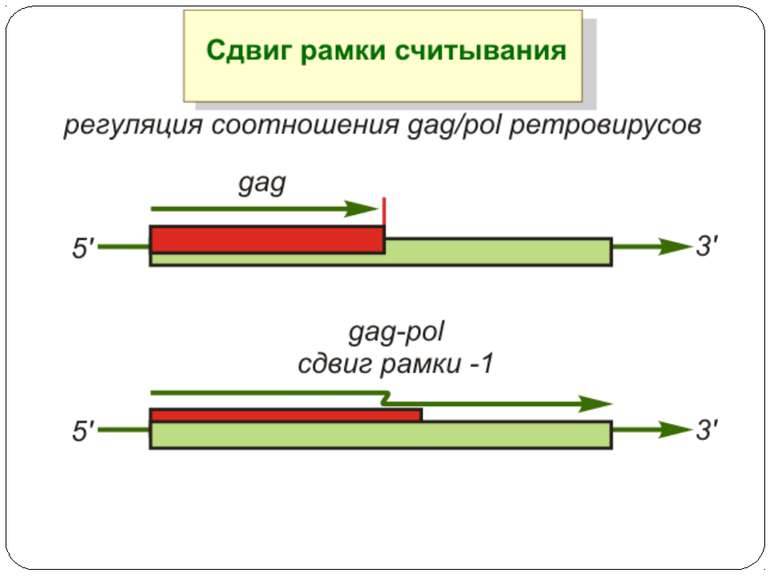
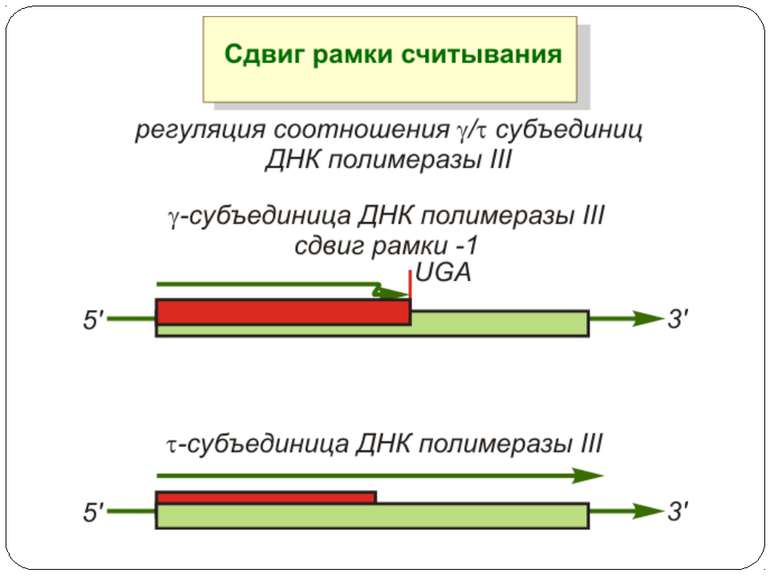
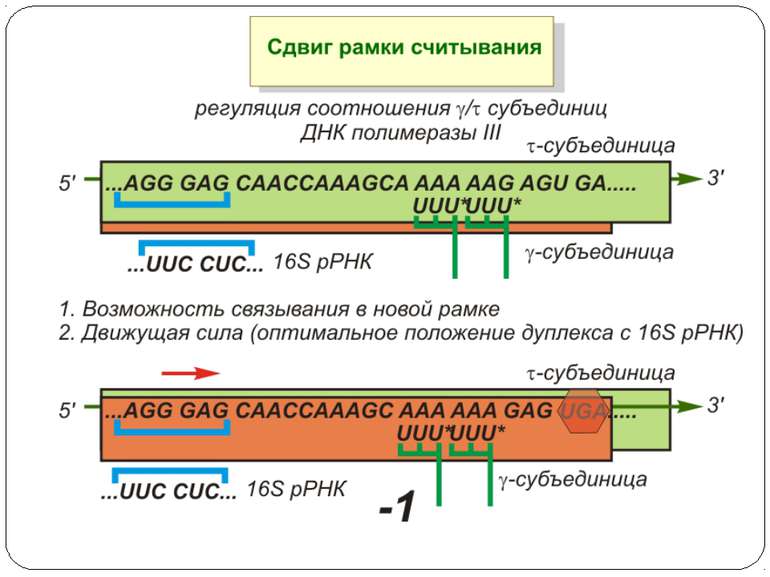
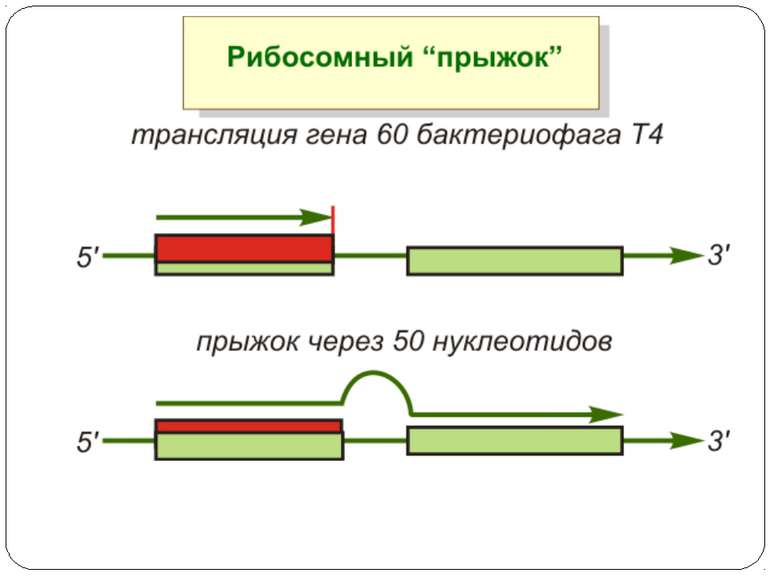
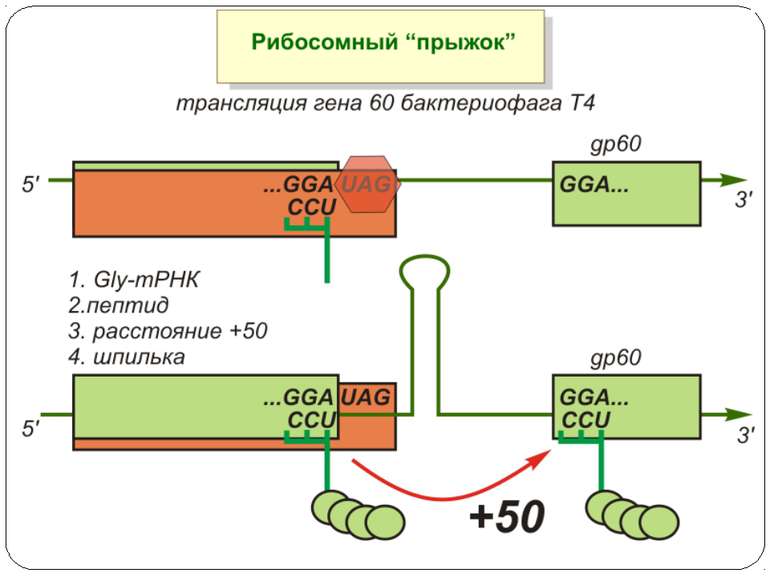
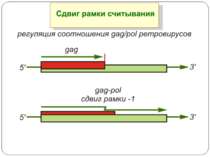
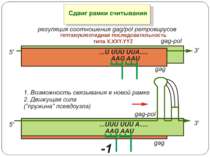
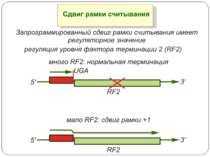
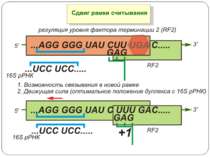
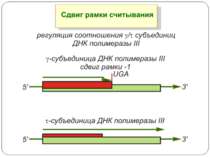
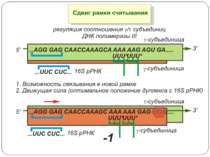
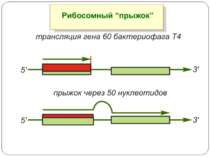
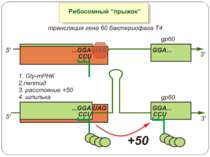
Регуляція елонгації Регуляція швидкості елонгації – зміна набору тРНК Зсув рамки зчитування Рибосомний “стрибок”
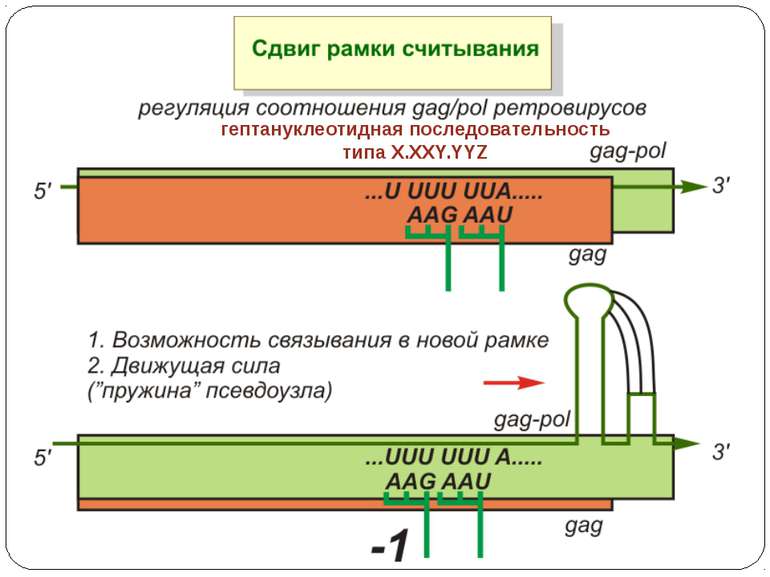
Сигнал до зсуву рамки зчитування у ретровірусів і ретротранспозонів Наявна гептануклеотидна послідовність типу X.XXY.YYZ (розмічена у вигляді кодонів у рамці зчитування 0), а також нижче розташований регуляторний елемент, що утворює вторинну структуру у вигляді шпильки або псевдовузла Зсув рамки відбувається в момент, коли пептидил-тРНК, пов'язана з кодоном XXY у Р-ділянці рибосоми, і аміноацил-тРНК, взаємодіюча з кодоном YYZ в А-Ділянці, одночасно зсуваються на один нуклеотид назад і стають напроти кодонів XXX і YYY трансльованої РНК. необхідно, щоб обидві молекули тРНК в А- і Р-ділянках утворювали міцний зв'язок з новими кодонами, які відрізняються від перших тільки нуклеотидами в положенні 3, що допускає неоднозначну відповідність антикодону Процес зсуву рамки викликається або підсилюється структурним елементом РНК, перед яким працююча рибосома робить паузу в трансляції
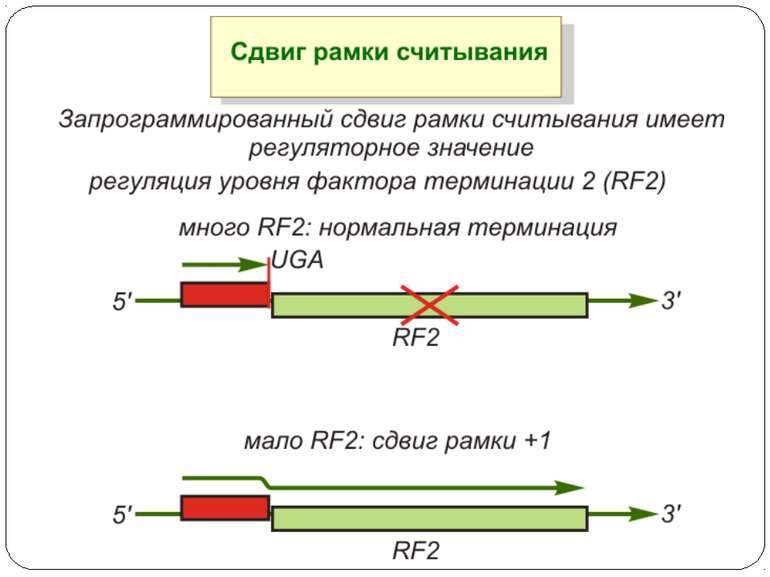
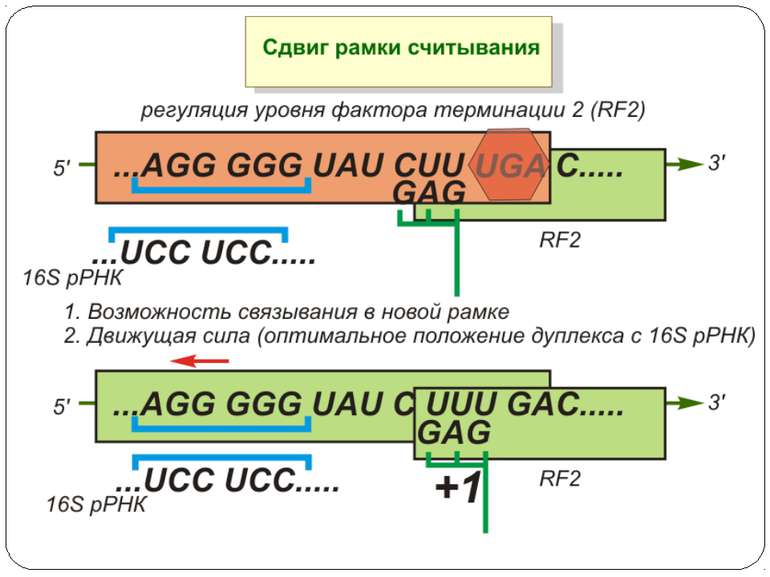
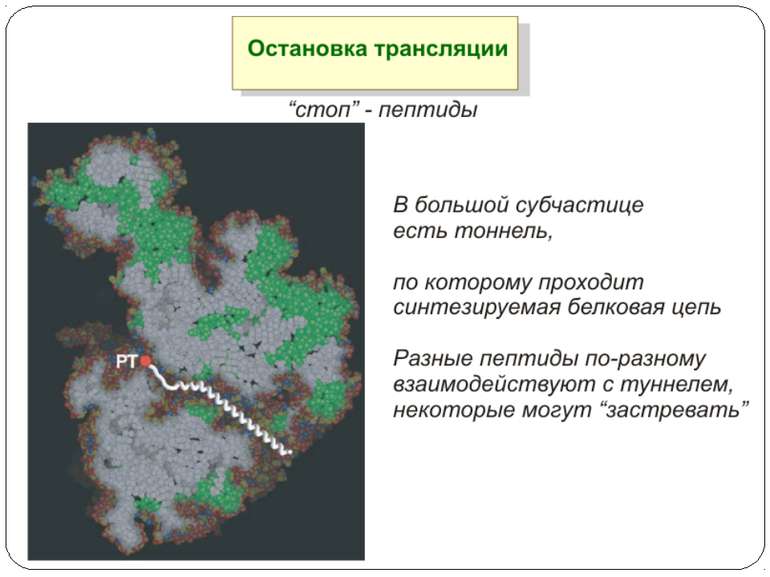
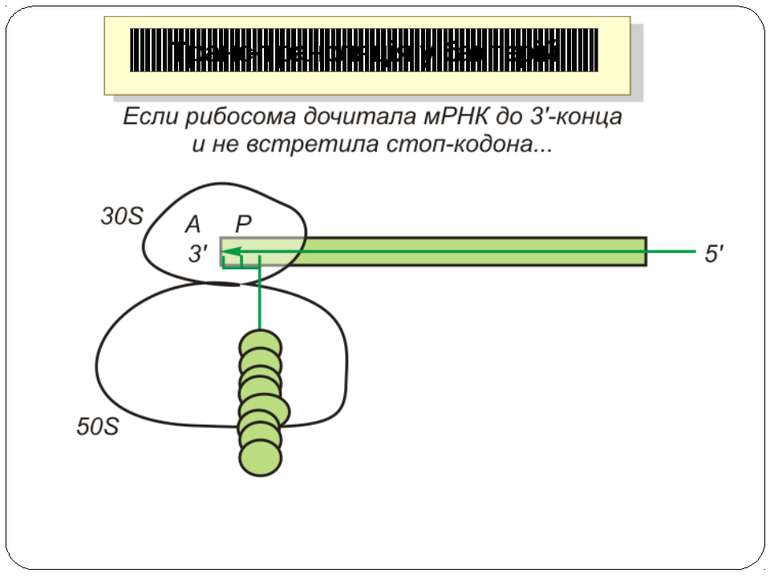
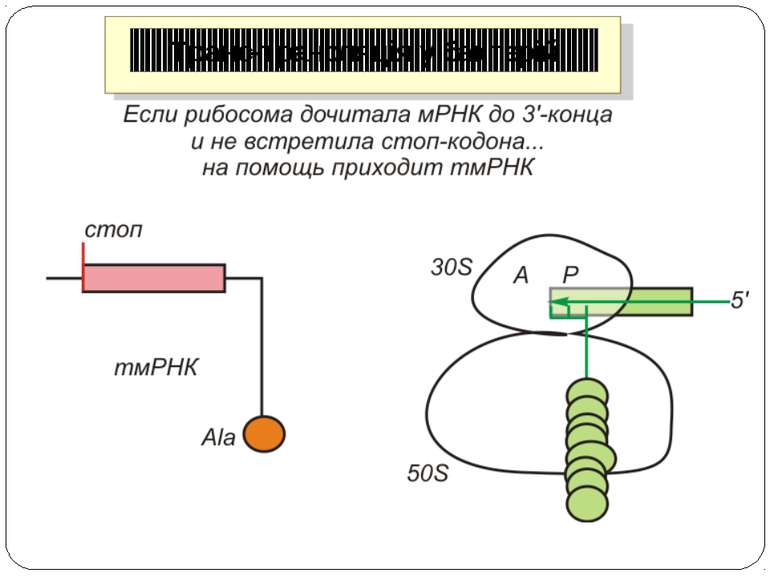
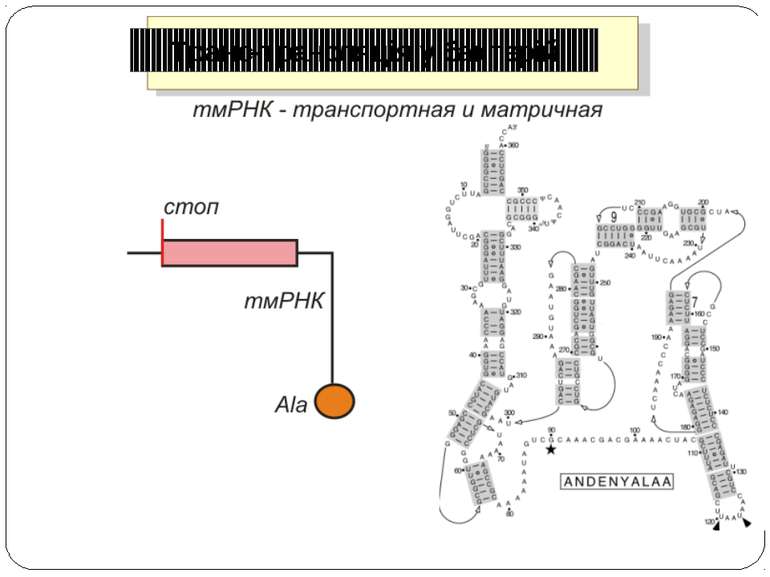
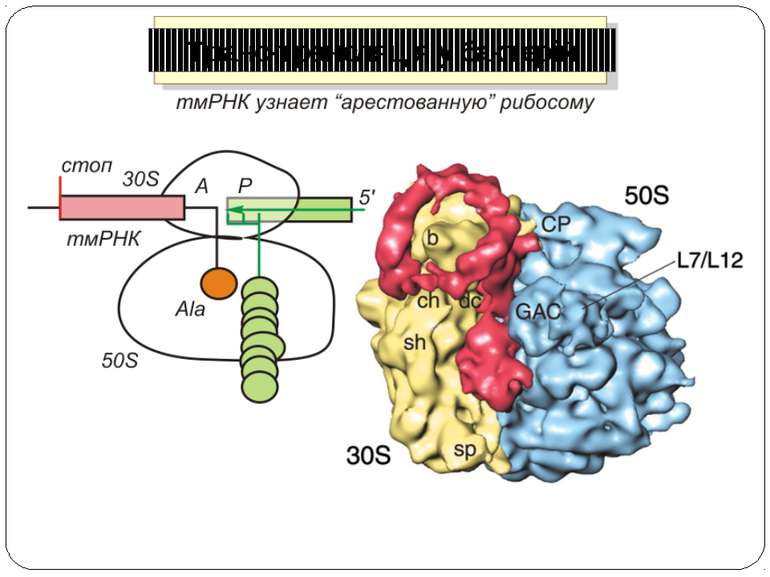
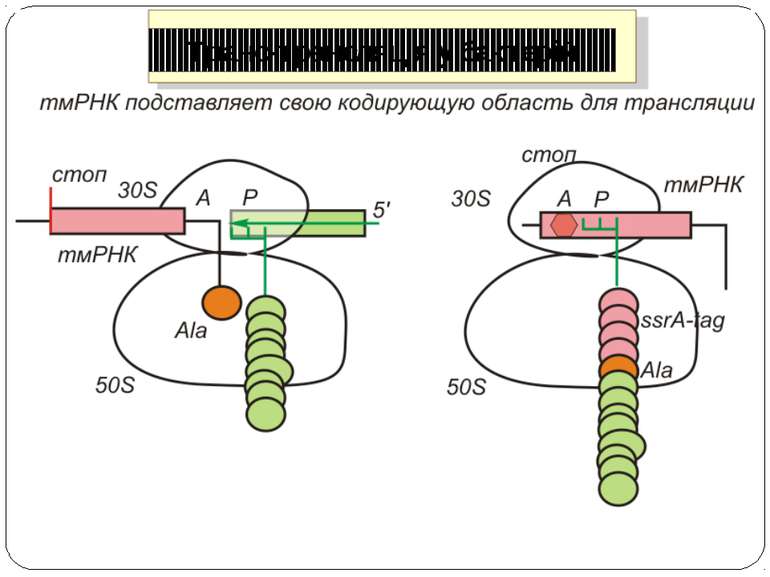
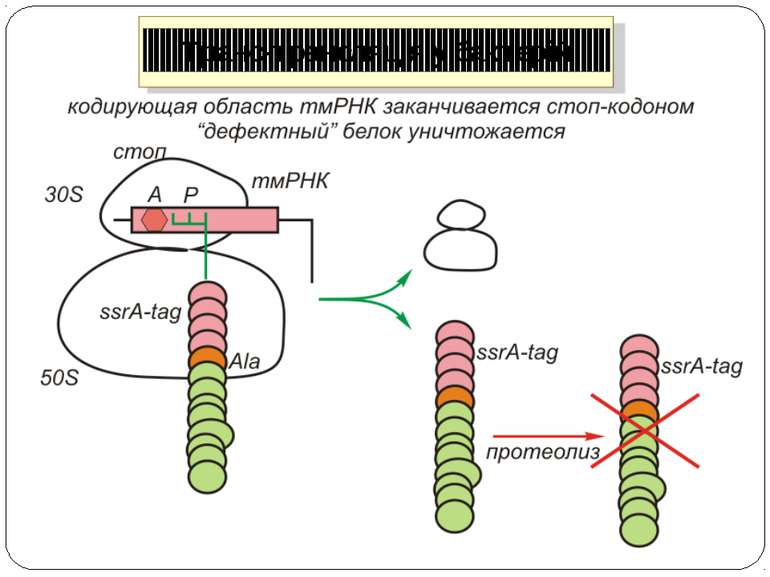
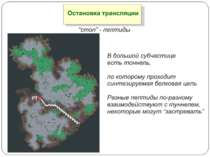
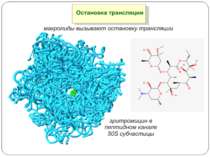
Регуляція на рівні термінації Рибосомний тунель як регулятор трансляції – арест трансляції (стоп-пептиди) Неправильне прочитання кодонів-термінаторів Транс-трансляція
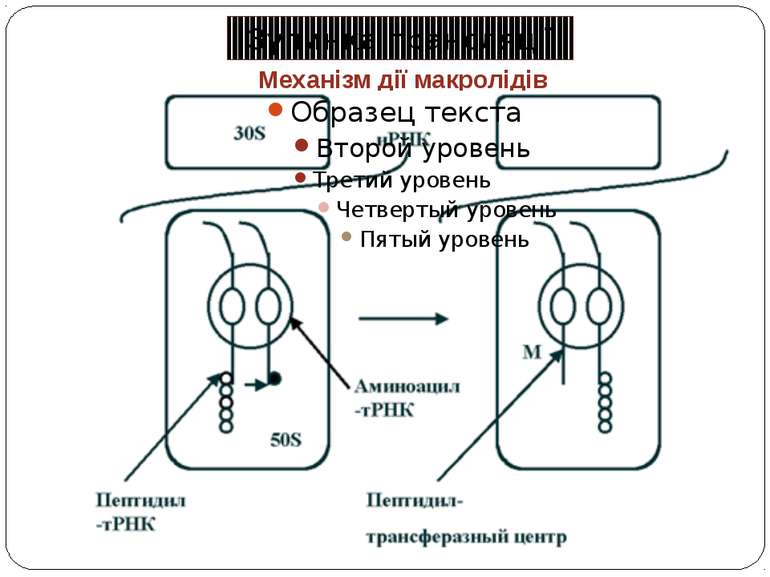
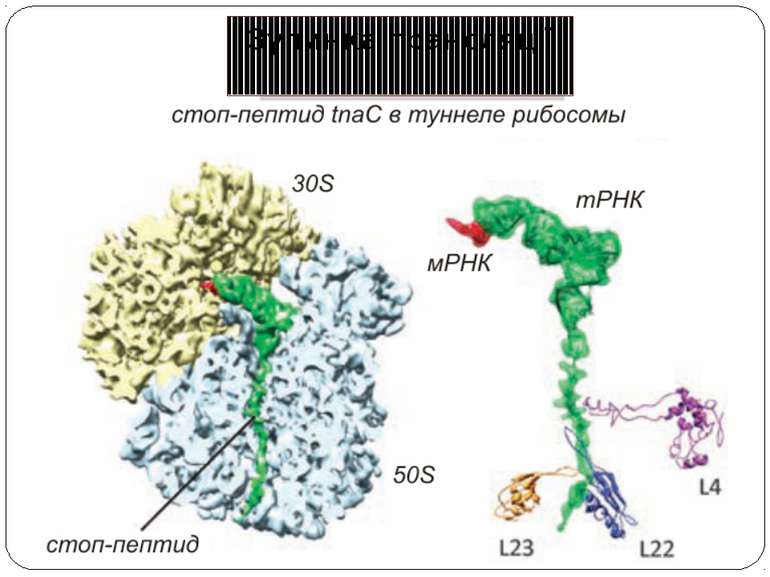
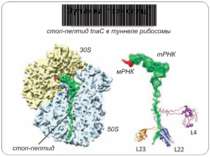
Рибосомний тунель як регулятор трансляції У РТ розташовані сайти зв'язування багатьох (у т.ч. клінічно важливих) антибіотиків і, як наслідок цього, модифікація стінок РТ приводить до стійкості бактерій стінки РТ беруть участь у моніторингу амінокислотної послідовності поліпептиду, що переміщається уздовж них зупинка трансляції лежить в основі механізмів регуляції експресії деяких генів, у тому числі відповідальних за секрецію, амінокислотний метаболізм, стійкість до антибіотиків
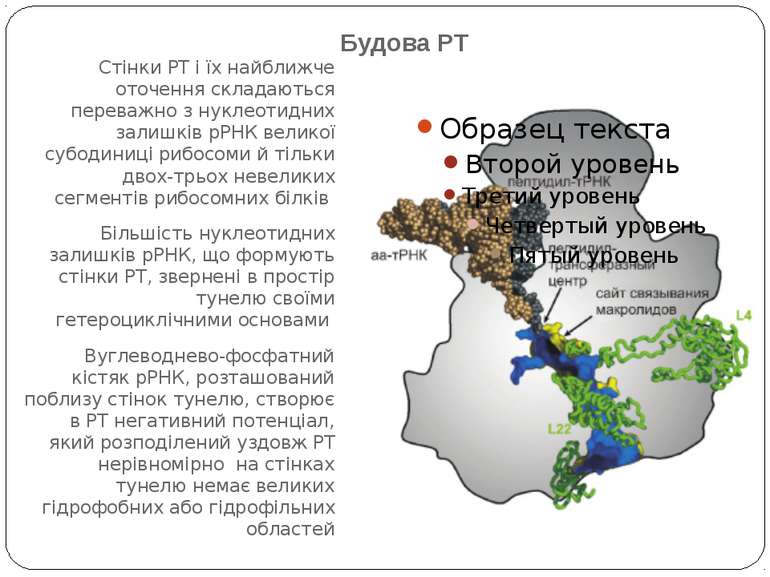
Будова РТ Стінки РТ і їх найближче оточення складаються переважно з нуклеотидних залишків рРНК великої субодиниці рибосоми й тільки двох-трьох невеликих сегментів рибосомних білків Більшість нуклеотидних залишків рРНК, що формують стінки РТ, звернені в простір тунелю своїми гетероциклічними основами Вуглеводнево-фосфатний кістяк рРНК, розташований поблизу стінок тунелю, створює в РТ негативний потенціал, який розподілений уздовж РТ нерівномірно на стінках тунелю немає великих гідрофобних або гідрофільних областей
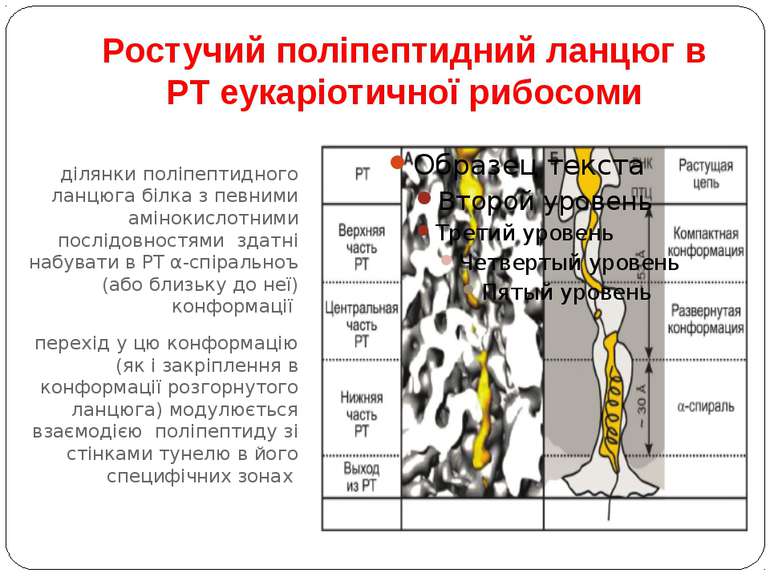
Ростучий поліпептидний ланцюг в РТ еукаріотичної рибосоми ділянки поліпептидного ланцюга білка з певними амінокислотними послідовностями здатні набувати в РТ α-спіральноъ (або близьку до неї) конформації перехід у цю конформацію (як і закріплення в конформації розгорнутого ланцюга) модулюється взаємодією поліпептиду зі стінками тунелю в його специфічних зонах
Рибосомний тунель як регулятор трансляції роль «стоп-пептидів» виконують т.зв. лідерні поліпептиди, закодовані в 5'-кінцевому районі оперону. зупинка їх трансляції в РТ регулює експресію генів, розташованих «униз за течією» (down-stream) відносно лідерної послідовності.
Рибосомний тунель як регулятор трансляції У генах, експресія яких регулюється через зупинку трансляції, закодовані мРНК, що володіють унікальною вторинною структурою, яка перебудовується так, як цього й вимагає класичний механізм аттенюації транскрипції. Н.: сайт зв'язування з рибосомою для послідовності, що кодує стійкість до лікарських препаратів, перебуває усередині однієї із двох інвертованих повторюваних послідовностей. Отже, у транскриптах цей сайт буде включений у стабільну структуру шпильки, яка перешкоджає трансляції. Лідерна послідовність, що кодує короткий регуляторний пептид, розташована «вище» (up-stream) цієї структури
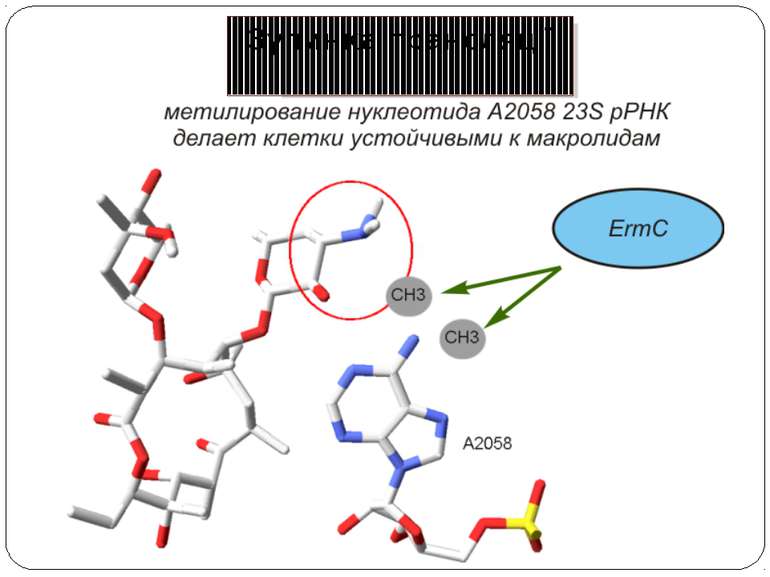
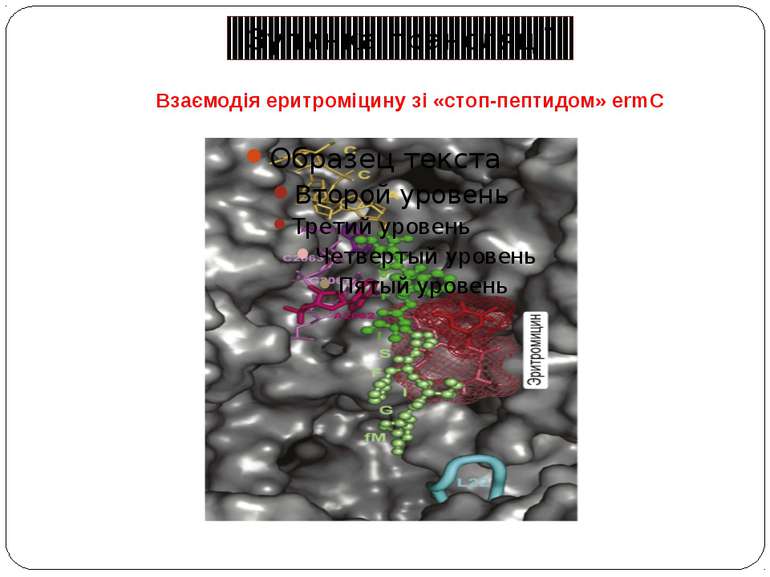
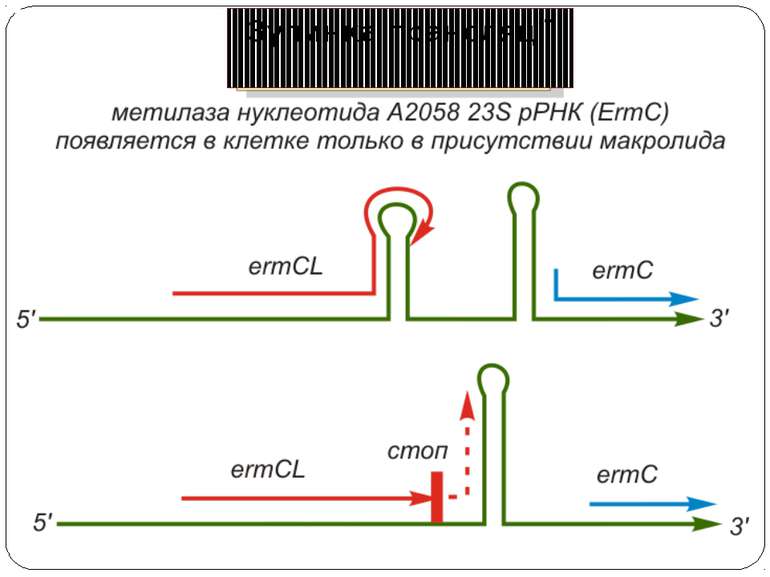
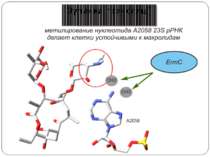
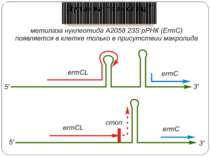
«Стоп-пептиди» інгібують трансляцію в комбінації зі специфічними низькомолекулярними коефекторами Основний етап аттенюації транскрипції – тимчасова зупинка рибосоми – викликаний інгібуванням пептидилтрансферазної активності короткими пептидами (5–8 амінокислотних залишків), що представляють собою N-кінцеві частини поліпептидів, закодованих у лідируючій послідовності регульованих генів. В Staphylococcus aureus експресія цього гена регулюється «стоп-пептидом», закодованим у лідерній послідовності (19 кодонів), розташованій на відстані 65 нуклеотидних залишків перед початком ermc. Під час відсутності антибіотиків відбувається конститутивна трансляція лідерної послідовності. При цьому мРНК білка Ermc не транслюється, тому що сайт зв'язування з рибосомою екранований у її вторинній структурі. У присутності антибіотика синтез лідерного пептиду зупиняється на 9-ом кодонові, вторинна структура мРНК перебудовується таким чином, що сайт ініціації трансляції білка Ermc стає доступним для рибосоми, і його синтез протікає безперешкодно .
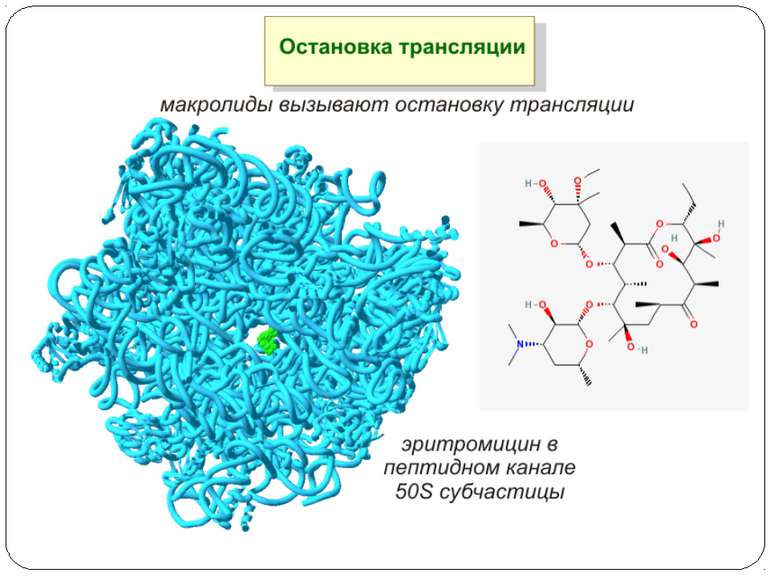
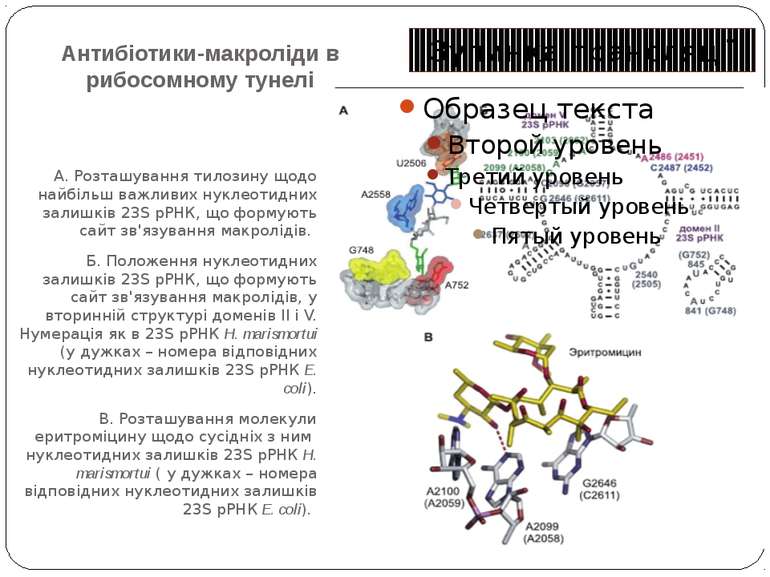
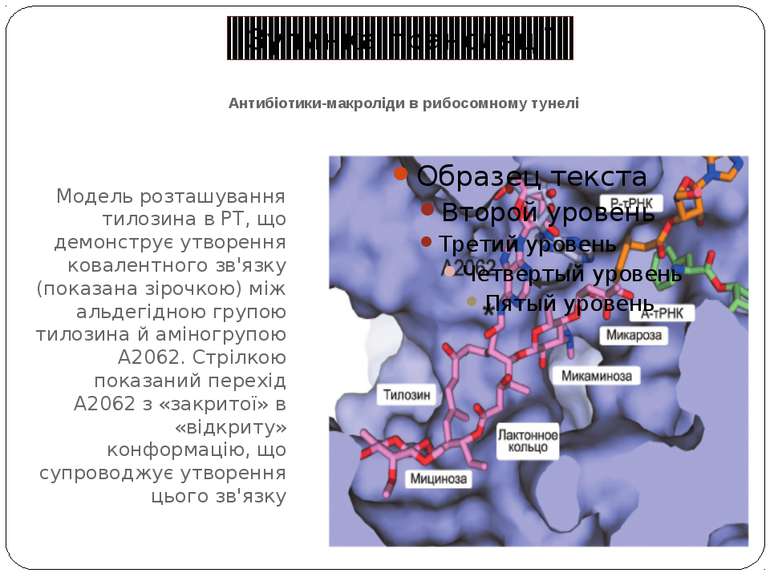
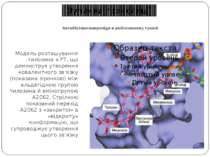
Антибіотики-макроліди в рибосомному тунелі А. Розташування тилозину щодо найбільш важливих нуклеотидних залишків 23S рРНК, що формують сайт зв'язування макролідів. Б. Положення нуклеотидних залишків 23S рРНК, що формують сайт зв'язування макролідів, у вторинній структурі доменів II і V. Нумерація як в 23S рРНК Н. marismortui (у дужках – номера відповідних нуклеотидних залишків 23S рРНК E. coli). В. Розташування молекули еритроміцину щодо сусідніх з ним нуклеотидних залишків 23S рРНК Н. marismortui ( у дужках – номера відповідних нуклеотидних залишків 23S рРНК E. coli). Зупинка трансляції
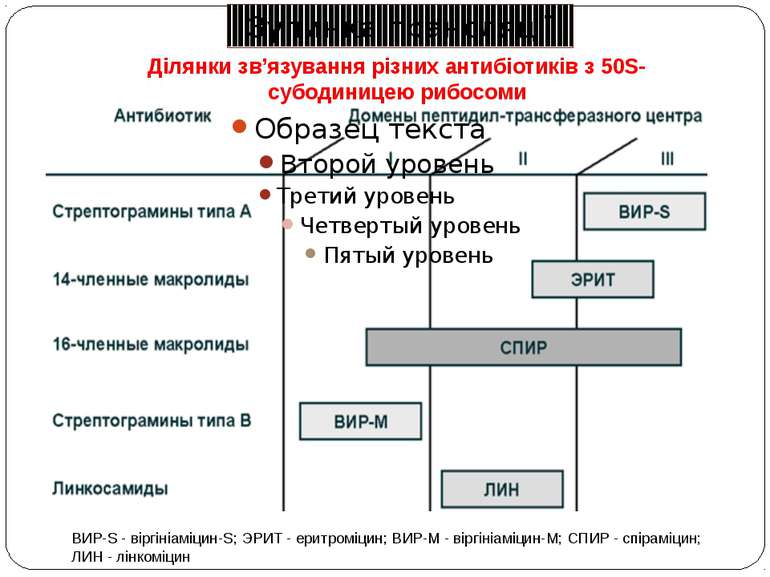
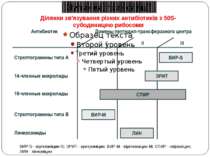
Ділянки зв’язування різних антибіотиків з 50S-субодиницею рибосоми ВИР-S - віргініаміцин-S; ЭРИТ - еритроміцин; ВИР-М - віргініаміцин-М; СПИР - спіраміцин; ЛИН - лінкоміцин Зупинка трансляції
Антибіотики-макроліди в рибосомному тунелі Модель розташування тилозина в РТ, що демонструє утворення ковалентного зв'язку (показана зірочкою) між альдегідною групою тилозина й аміногрупою А2062. Стрілкою показаний перехід А2062 з «закритої» в «відкриту» конформацію, що супроводжує утворення цього зв'язку Зупинка трансляції
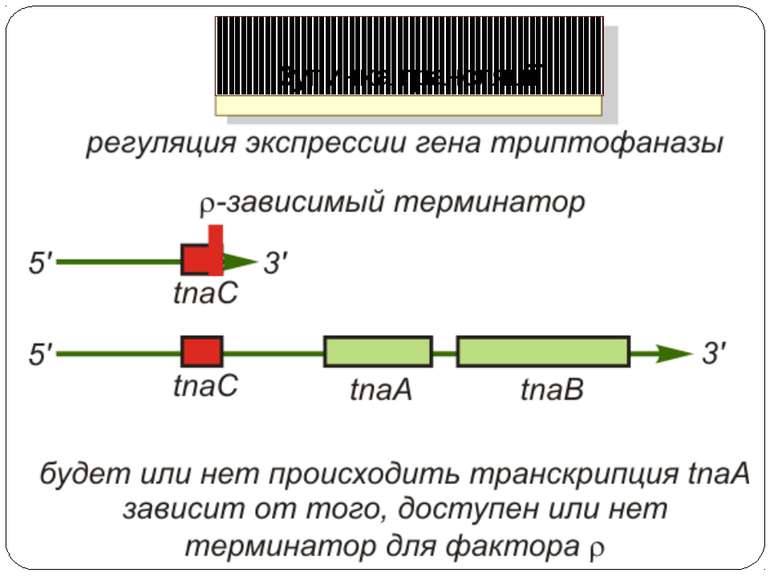
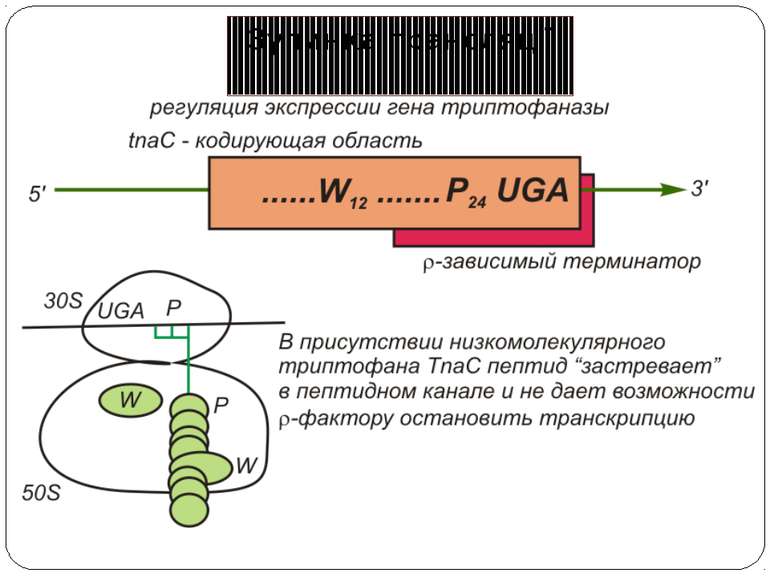
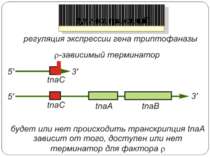
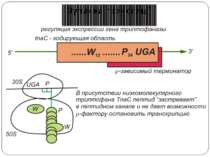
Участь лідерного пептиду tnaС у регуляції триптофаназного оперона Триптофаназный (tnА) оперон E. coli складається із лідерної регуляторної послідовності (320 п.о.), за якою ідуть два структурні гени tnaА і tnaB. У лідерній послідовності закодований 24-нуклеотидний регуляторний –«стоп-пептид» TnaС; tnaА кодує триптофаназу, а tnaB – триптофан-пермеазу. Зупинка трансляції
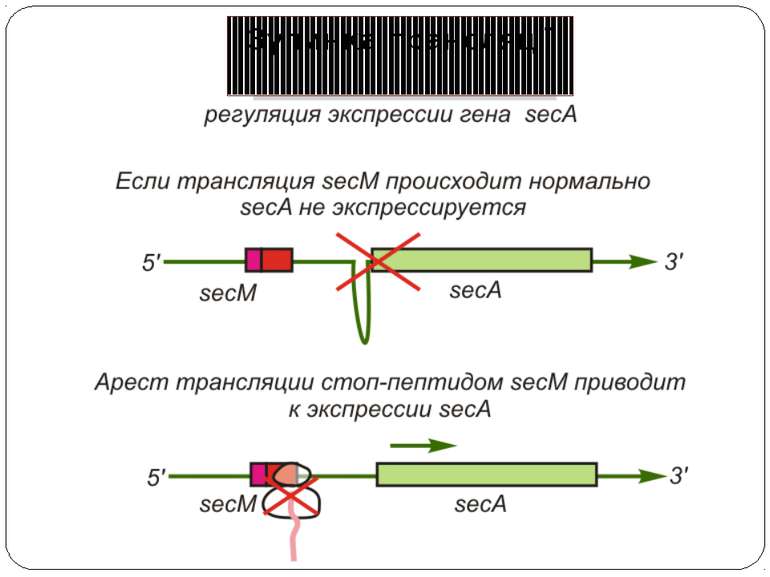
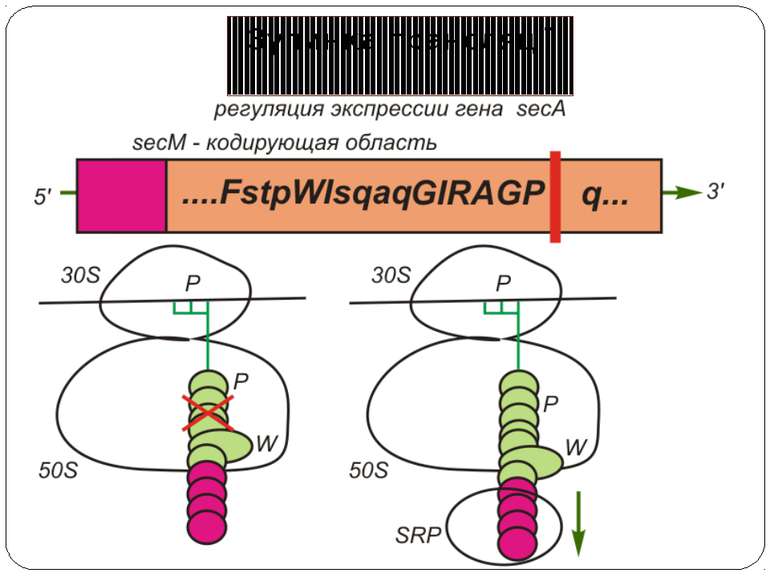
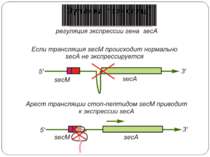
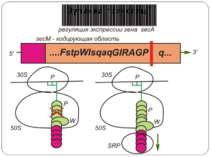
Білок SecА відіграє важливу роль у секреції й транспорті через мембрани білків E. сoli Експресія гена secА в E. coli регулюється його лідерною ділянкою, що містить відкриту рамку зчитування (ОРС), у якій закодований білок secМ (170 кодонів). Коли на лідерній мРНК сайт зв'язування рибосоми доступний, подібний сайт на ділянці secА залучений в утворення вторинної структури мРНК, що перешкоджає ініціації синтезу SecА. Відбувається ініціація трансляції й полімеризація перших 165 амінокислот доти, поки в Р-сайт не потрапить Gly-Трнк, відповідна до кодону Gly165, на якому рибосома зупиняється. Поліпептидний ланцюг SecМ містить сигнальну послідовність на N-кінці, і якщо апарат секреції білків у клітині працює нормально, те N-кінцева частина пептиду SecМ буде виведена з тунелю, що спричинить вивільнення рибосоми з арештованого стану за допомогою SRP. У цьому випадку активації експресії гена secА не відбувається. Ключовий момент роботи цієї системи полягає в тому, що арешт рибосоми відбувається при нестачі SecА у клітині, тобто в умовах порушення секреції білків. Рибосома зупиняється на 165 кодону secМ, у результаті чого вторинна структура мРНК перебудовується,і інша рибосома здійснює трансляцію тієї її частини, у якій закодований білок Seca
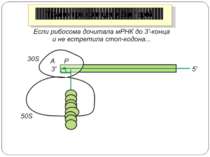
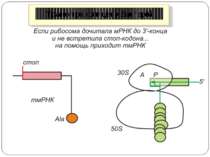
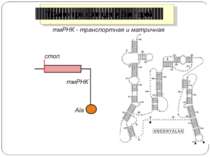
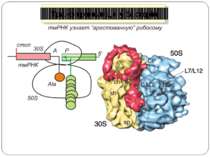
Неповна термінація синтезу поліпептидів UGA Він частіше інших помилково розпізнається транслюючою рибосомою як осмислений (очевидно, за участю триптофанової тРНК). У результаті синтезується більш довгий поліпептид, припинення синтезу якого відбувається на наступному термінуючому кодоні. Н.: при трансляції РНК фага Q-бета. Цистрон білка оболонки фага закінчується термінуючим кодоном UGA, який з невеликою частотою розпізнається рибосомами як осмислений, що приводить до синтезу більш довгого, чим білок оболонки, поліпептиду. Цей поліпептид потрібен для збирання повноцінної (життєздатної) фагової частки і є життєво важливим для бактеріофага Q-бета. Зупинка трансляції
UAG Для утворення гібридного білка Gag-Pol ретровіруси типу С використовують супресію термінуючого кодону замість зсуву рамки зчитування. Супресія відбувається з ефективністю близько 5% і супроводжується помилковим прочитуванням UAG-кодону глутамініл-тРНК. Термінуючий кодон UGA у тому ж положенні декодується як аргініновий, цистеїновий або триптофановий. Звичайні термінуючі кодони нормальних клітинних генів у цих умовах не супресуються, тому для здійснення супресії термінуючий кодон повинен перебувати в певному контексті. Зупинка трансляції Неповна термінація синтезу поліпептидів
Трансляційні енхансери Специфічні послідовності мРНК, розміщені в 5' и 3'- нетрансльованих областях поблизу послідовності Шайна-Дальгарно і кодону AUG , що сприяють відбору молекул мРНК рибосомою. Енхансерні послідовності впізнають комплементарні ділянки16S рРНК, з якою вони взаємодіють на стадії утворення ініціаторного контакту мРНК з рибосомою. Вважається, що ця взаємодія приводить до відносного збільшення концентрації мРНК поблизу 30S субодиниці рибосоми , підвищуючи таким чином можливість трансляції мРНК.
Енхансер “епсилон” UUAACUUUA Виявлений поблизу послідовності Шайна-Дальгарно в гені 10 фага Т4. Ця AU- багата послідовність значно посилює експресію даного гену, а також ефективно функціонує при розміщенні перед іншими неродинними генами. Схожий енхансерний элемент (UUAAUUUAC і UUAACU) виявлено в гені atp Escherichia coli. Послідовність «епсилон" комплементарна нуклеотидам 458-466 16S рРНК E. coli , що розміщені на поверхні субодиниці рибосомы. Вважається, що ці пірімідин-багаті послідовності можуть також взаємодіяти з рибосомним білком S1.
Энхансер трансляционный омега Энхансер трансляции "омега" был обнаружен в 5'- некодирующей области РНК вируса табачной мозаики. Эта последовательность из 68 нуклеотидов усиливает трансляцию как в прокариотических, так и в эукариотических трансляционных системах in vivo и in vitro. Последовательность "омега" обогащена AU и не комплементарна области 460 в 16S рРНК , как последовательность "эпсилон" . В тех случаях, когда в матричной РНК отсутствует последовательность Шайна-Дальгарно , последовательность "омега" функционально эквивалентна ей и способна усиливать трансляцию в 40-120 раз, что тем не менее в 10 раз ниже уровня трансляции нормальной мРНК. В присутствии последовательности Шайна-Дальгарно последовательность "омега" усиливает трансляцию до 4 раз. Последовательность "омега" располагается на мРНК в районе до - 110 нуклеотида от стартового сайта. Тандемные копии этой последовательности удваивают эффект усиления трансляции у E. coli. Наиболее важным элементом последовательности "омега" является последовательность ACAAUUAC, повторенная трижды. Предполагается, что пентануклеотид CAAUU комплементарно взаимодействует с последовательностью AAUUG, расположенной в 5'- области 16S рРНК. Эта область мРНК высококонсервативна у различных бактериальных видов и является потенциальным сайтом связывания трансляционных энхансеров. Енхансер “омега”
Схожі презентації
Категорії