Презентація на тему:
11_10__3_Регуляція транскрипції еукаріот 11 10 11
Завантажити презентацію
11_10__3_Регуляція транскрипції еукаріот 11 10 11
Завантажити презентаціюПрезентація по слайдам:
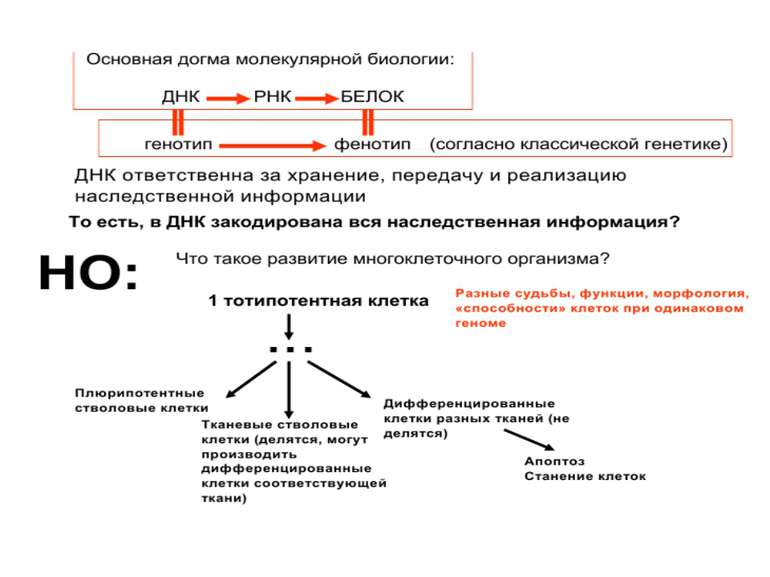
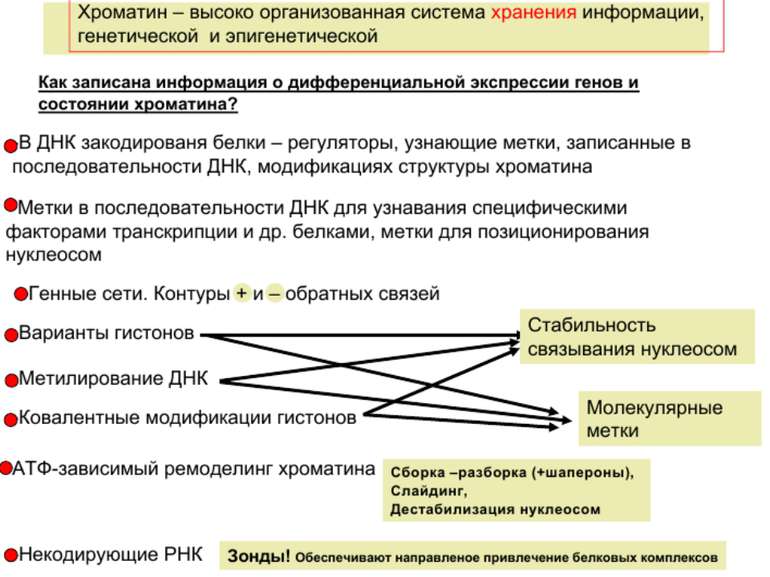
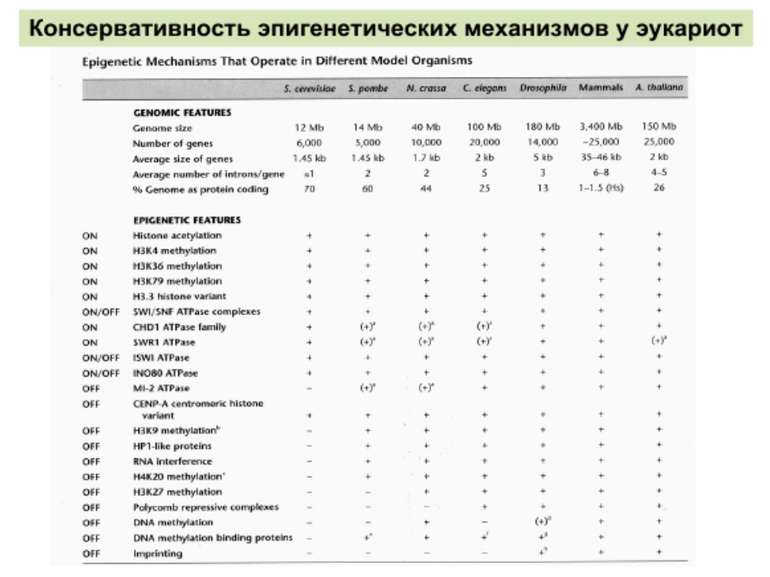
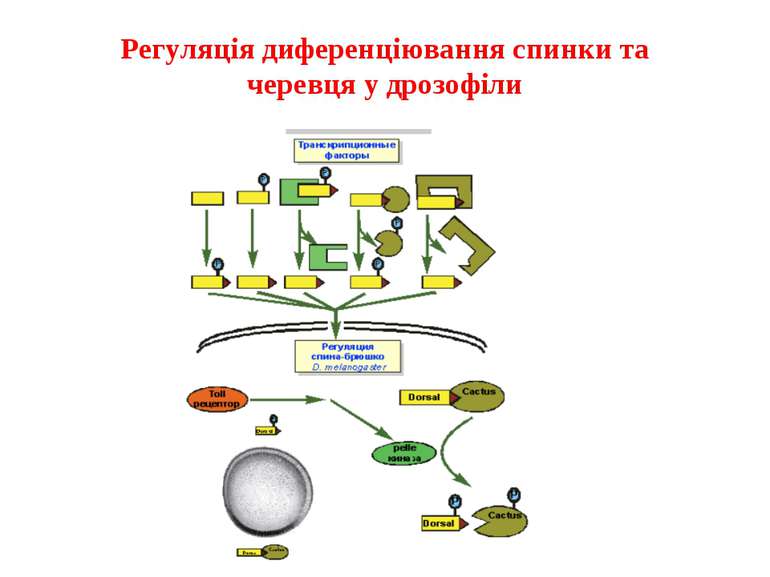
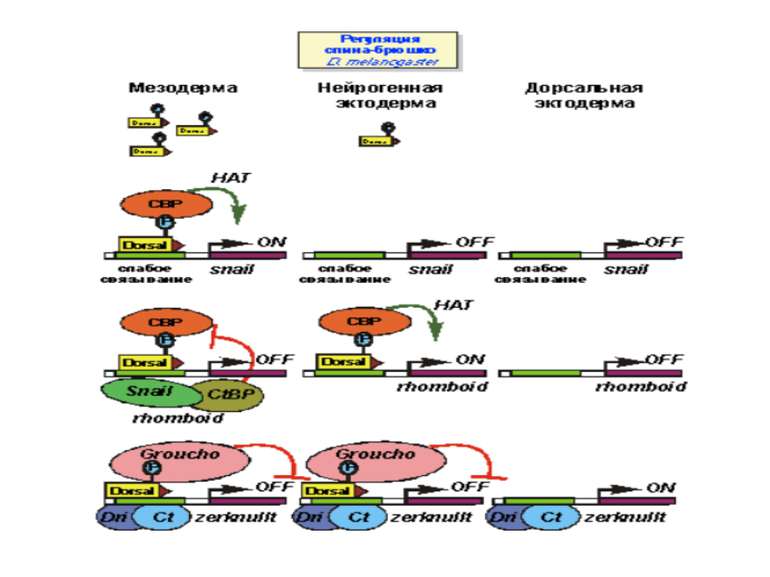
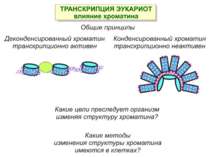
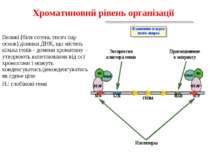
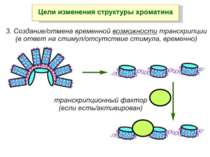
Регуляція експресії генів може здійснюватись на різних рівнях Хроматиновий рівень – конденсація хроматину та доменів хроматину Численні ділянки геному регулюються як єдині протяжні домени хроматину. На цьому рівні можлива епігенетична регуляція (епігенетичне успадкування стану експресії) Окремі гени У цій регуляцї беруть участь: Ковалентні модифікації ДНК Варіанти і модифікації гістонів Різні негістонові білки хроматину Просторова організація ядра Просторово-часова картина реплікації
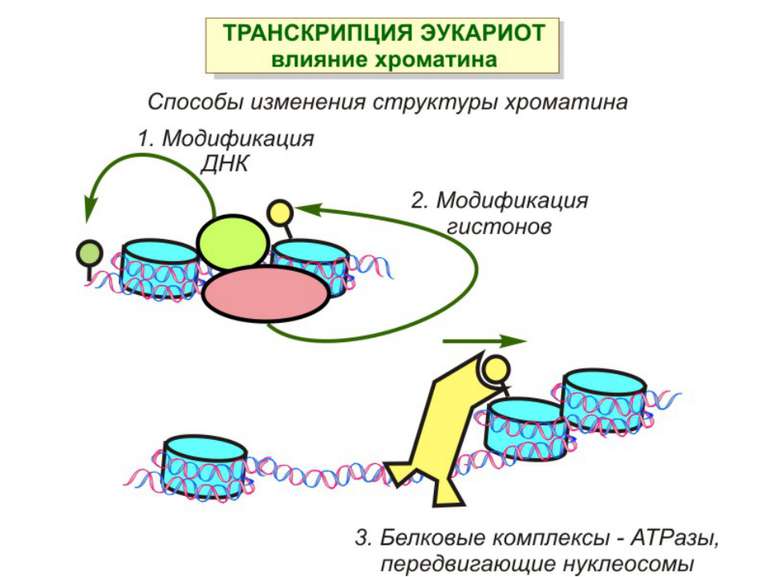
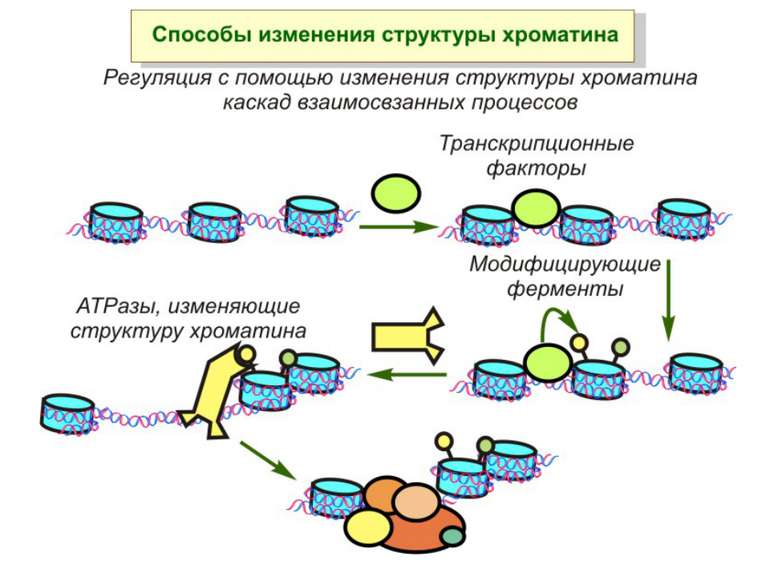
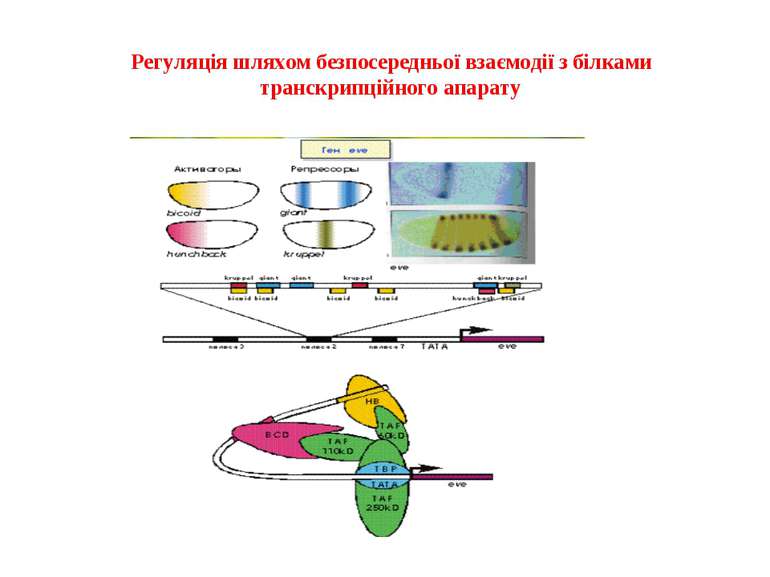
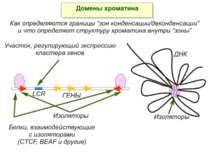
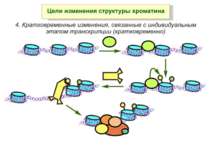
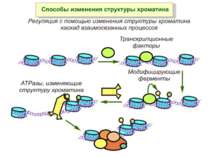
Механізми регуляції транскрипції еукаріот Зв’язування з ДНК регуляторних білків і факторів транскрипції. Модифікація ДНК (метилювання) і гістонів (ацетилювання, метилювання і ін.) Компактизація/декомпактизація хроматину, що здійснюється білковими комплексами
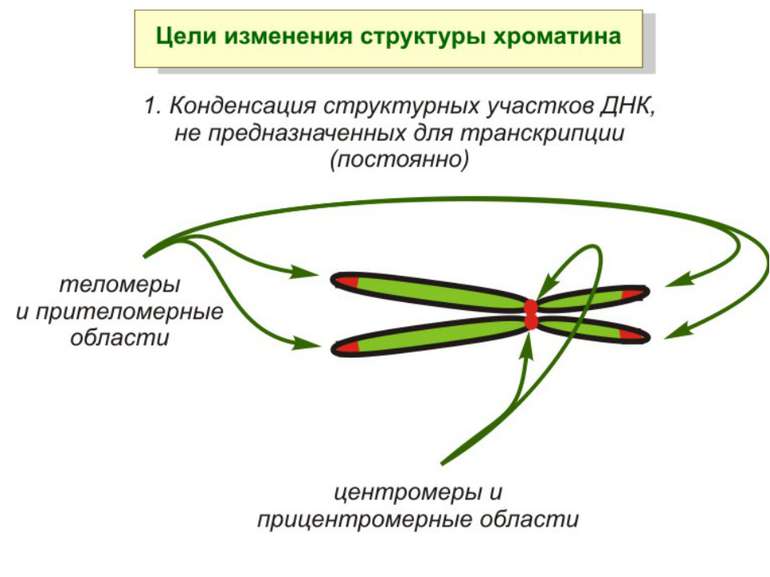
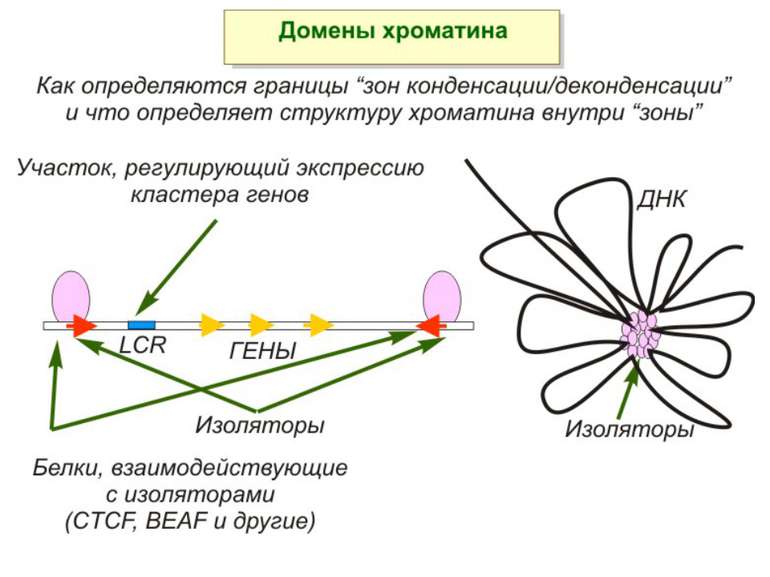
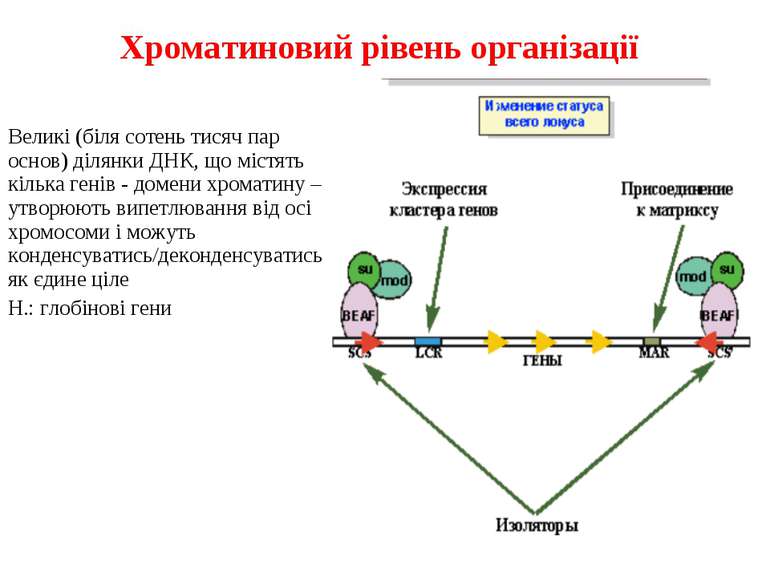
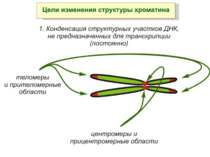
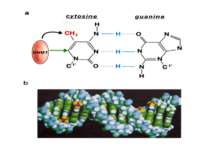
Хроматиновий рівень організації Великі (біля сотень тисяч пар основ) ділянки ДНК, що містять кілька генів - домени хроматину – утворюють випетлювання від осі хромосоми і можуть конденсуватись/деконденсуватись як єдине ціле Н.: глобінові гени
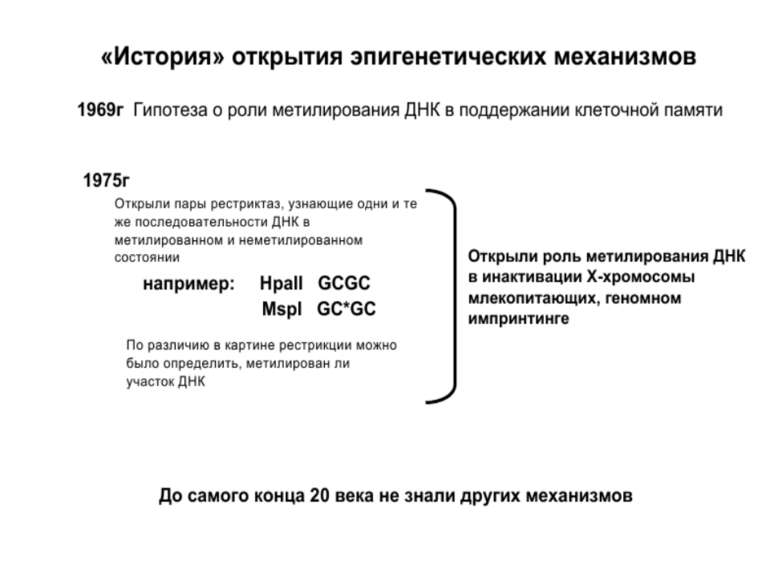
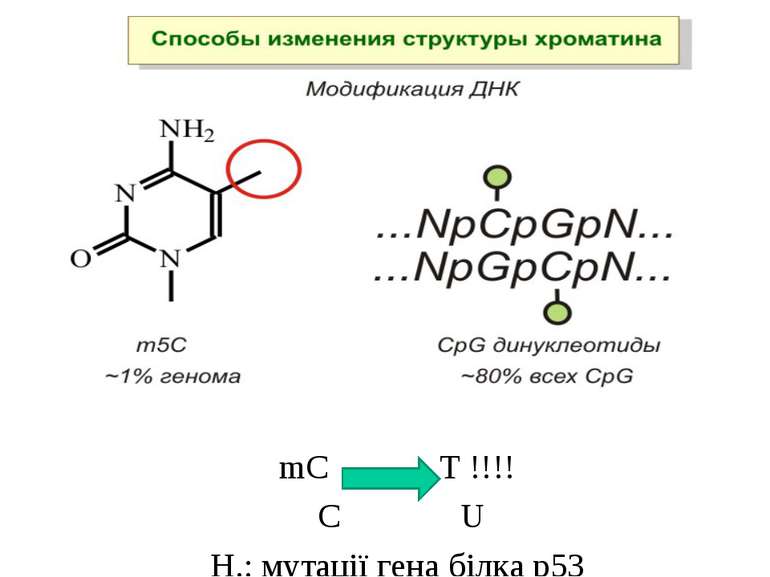
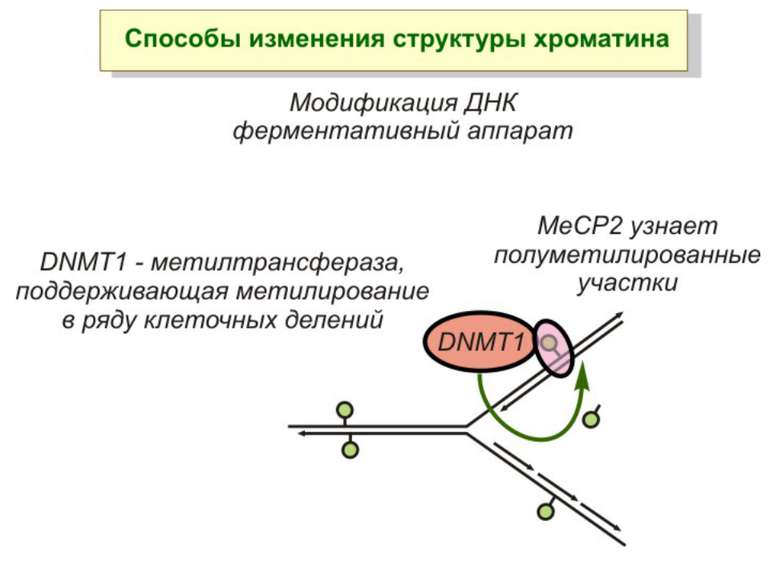
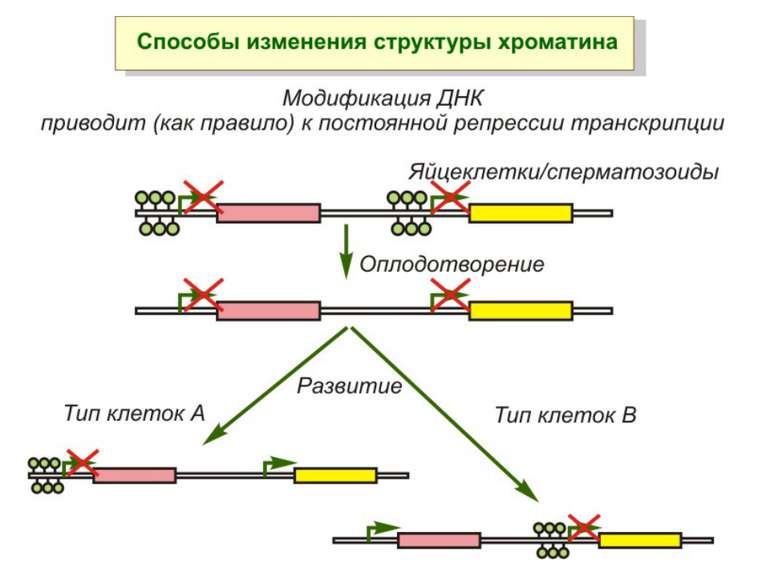
Роль метилювання ДНК в регуляції транскрипції Вибірковість метилювання Карти метилювання (батьківський імпрінтинг, хвилі онтогенезу) Метилювання і онкогенез Участь в інактивації Х-хромосоми (синдром Тернера)
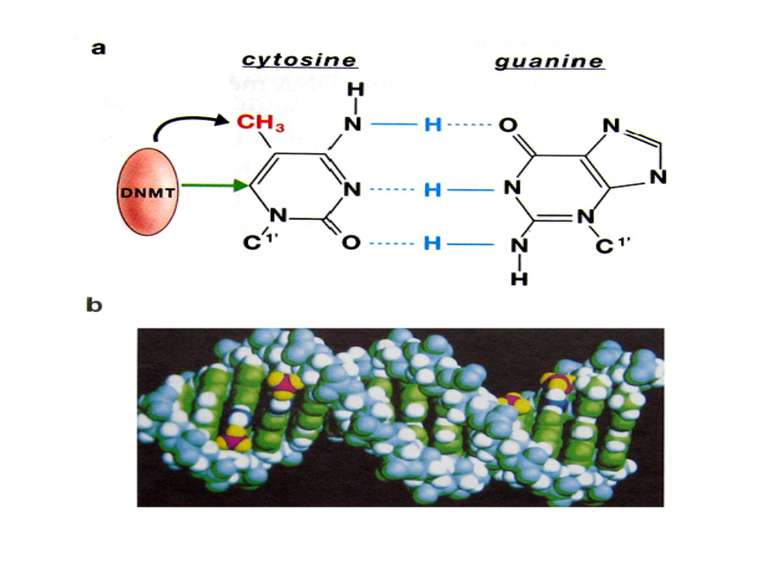
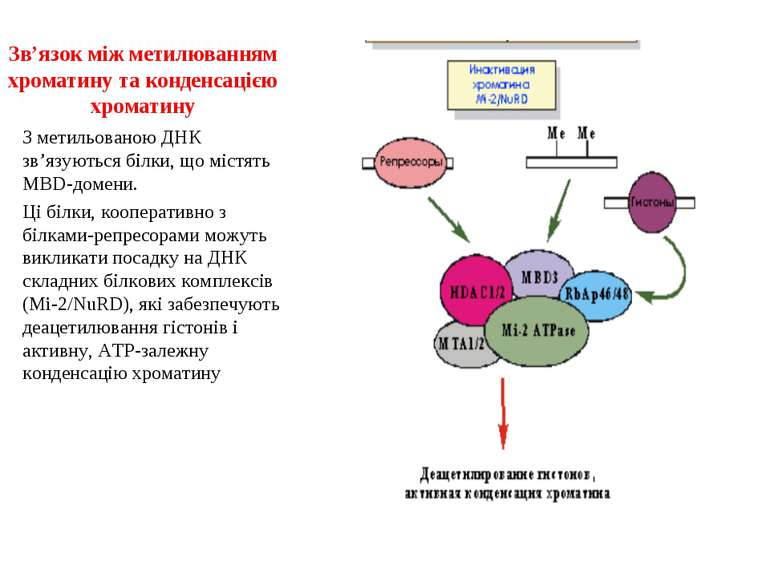
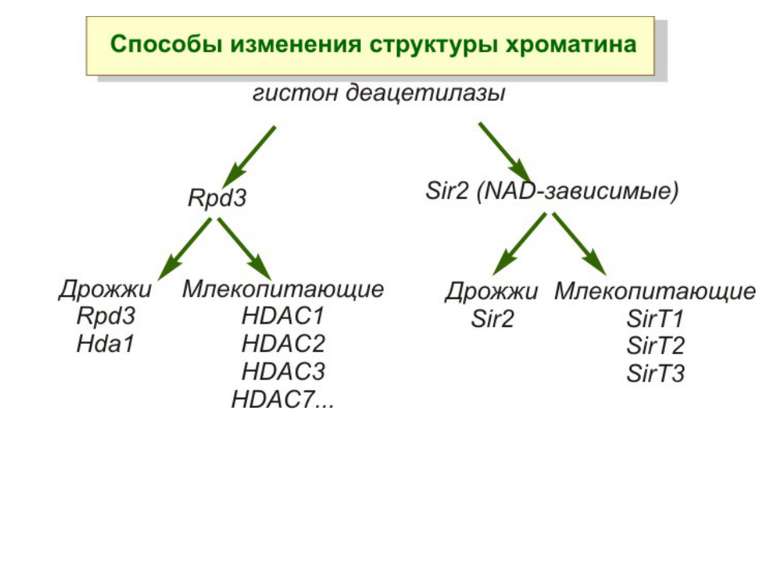
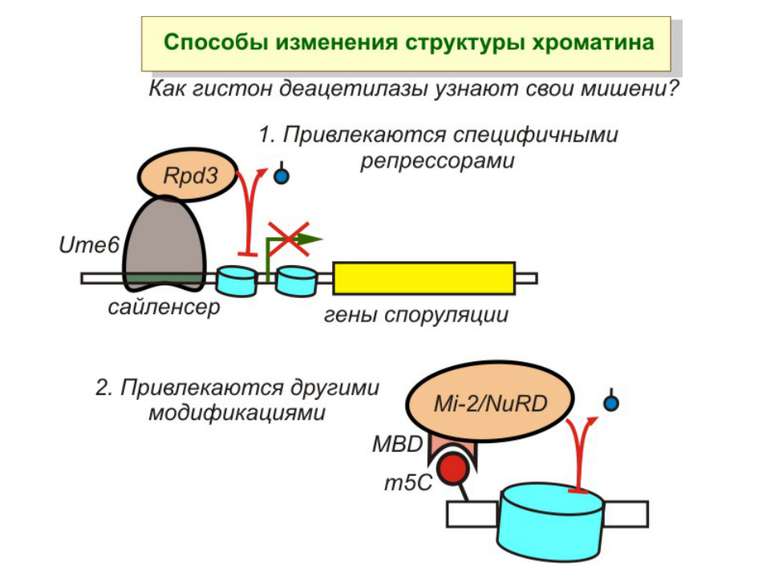
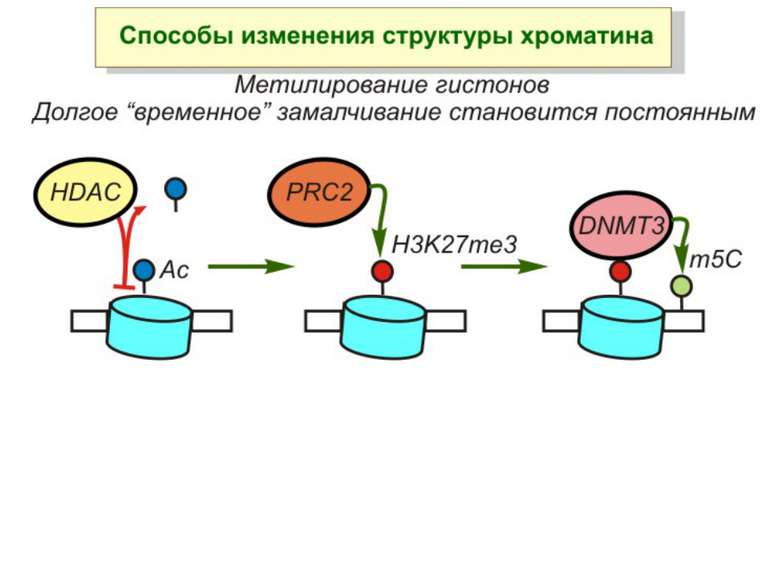
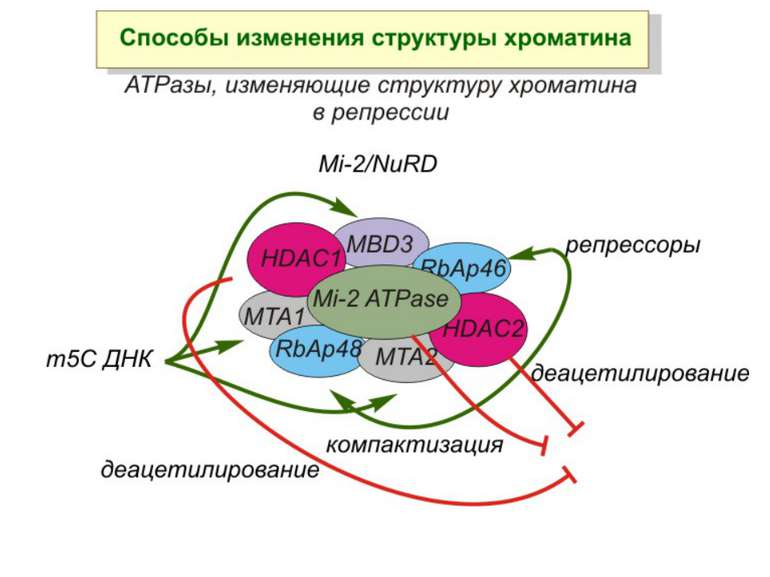
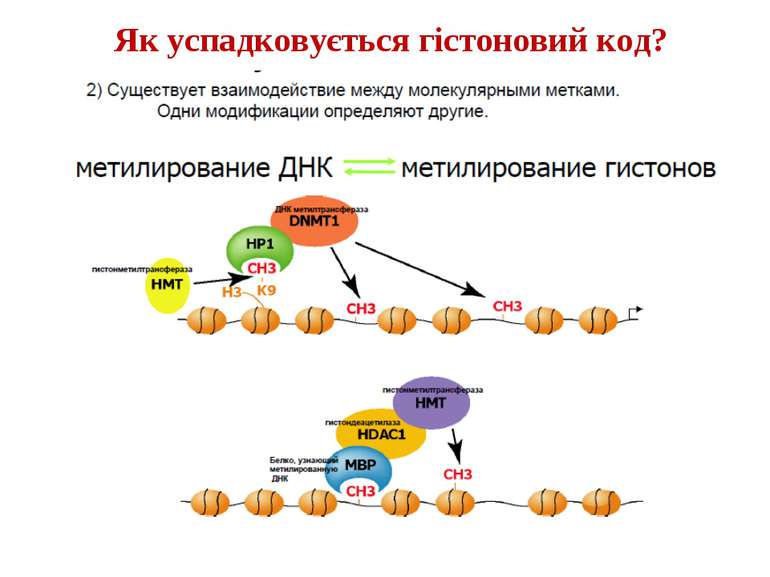
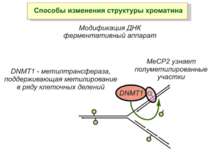
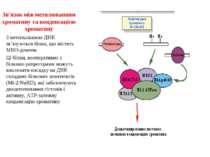
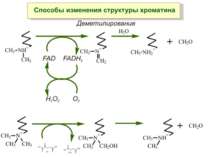
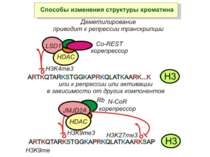
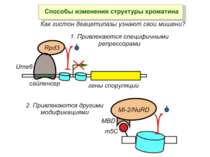
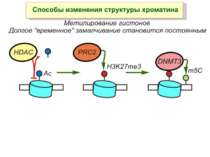
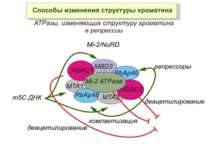
Зв’язок між метилюванням хроматину та конденсацією хроматину З метильованою ДНК зв’язуються білки, що містять MBD-домени. Ці білки, кооперативно з білками-репресорами можуть викликати посадку на ДНК складних білкових комплексів (Mi-2/NuRD), які забезпечують деацетилювання гістонів і активну, ATP-залежну конденсацію хроматину
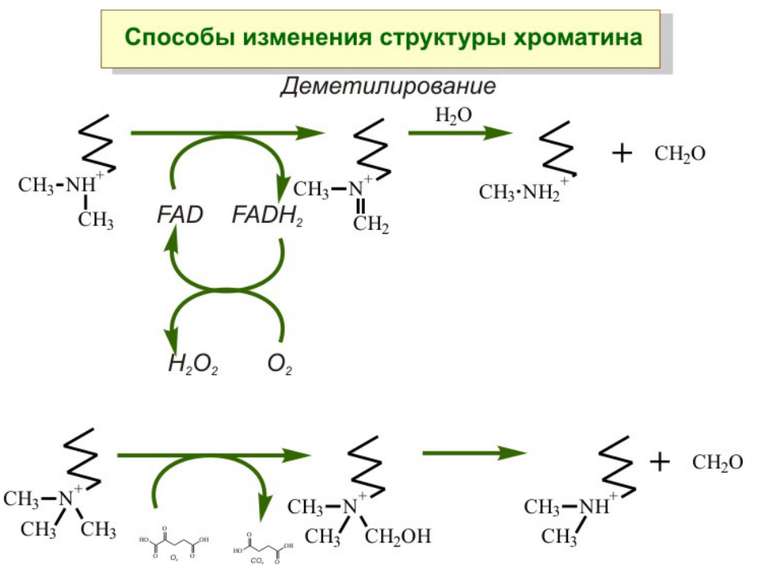
Передача карт метилювання Хвилі метилювання в онтогенезі (пасивне і активне деметилювання ((фактори транскрипції?))
Передача карт метилювання Батьківський імпритинг – сімейні спадкові захворювання аА* і аа Характерне для хребетних
Передача карт метилювання Еволюційне значення “Конфлікт інтересів” Ген фактору росту плаценти активний, якщо успадковується від батька, і гіперметильований (неактивний), якщо приходить від матері. Навпаки, ген, що інгібує фактор росту, активний лише в тому випадку, якщо успадковується від матері.
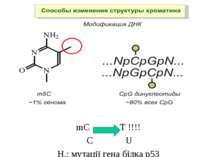
Метилювання і онкогенез Метилювання промоторів генів-супресорів Н.: р53 Rb (retinoblastoma)
Метилювання і інактивація Х-хромосоми Генний баланс – недопустимість дуплікацій аутосом Неповне виключення Випадковий вибір синдром Тернера – вплив походження Х-хромосоми
Метилювання аденінових залишків ДНК N6-метиладенін Залучене в регуляцію експресії генів і реплікацію, в тому числі і мітохондріальних ДНК. Може контролювати персистенцію чужерідних ДНК в клітині і, ймовірно, служить елементом системи рестрикції-модифікації у рослин.
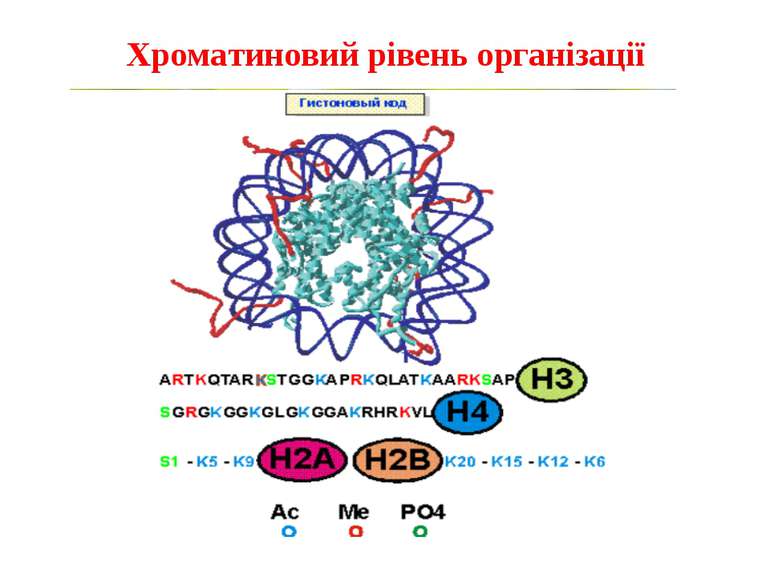
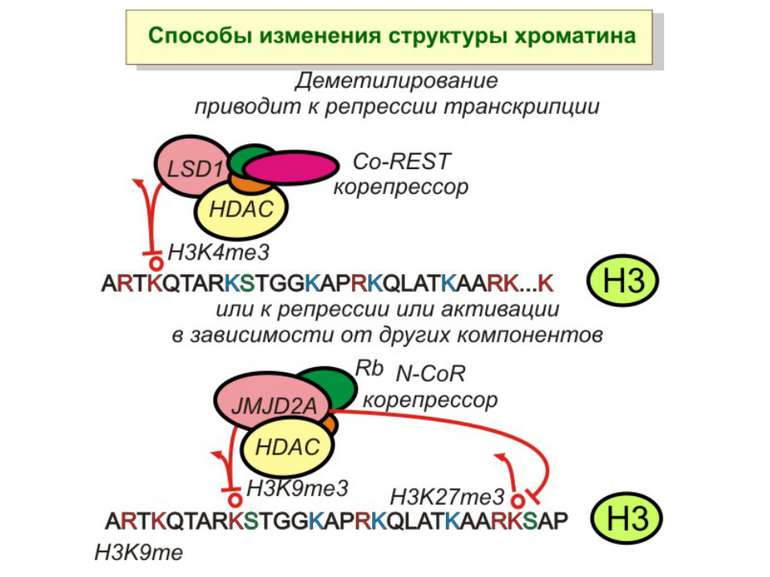
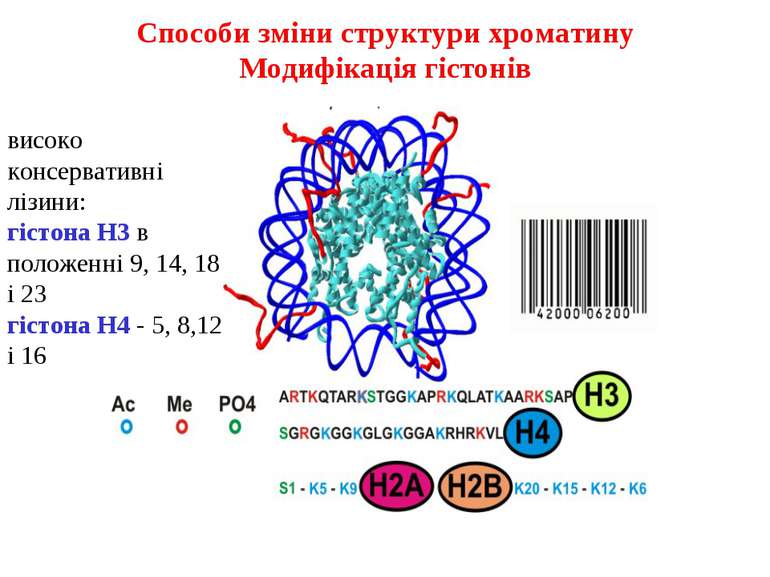
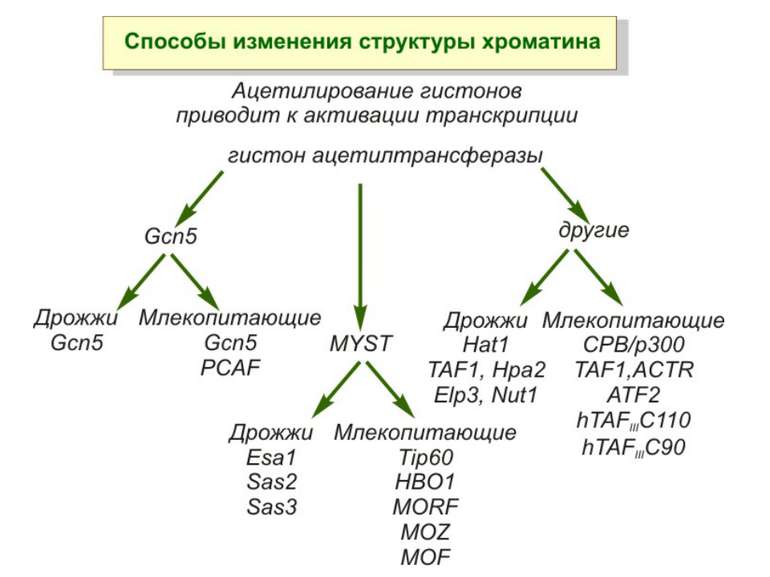
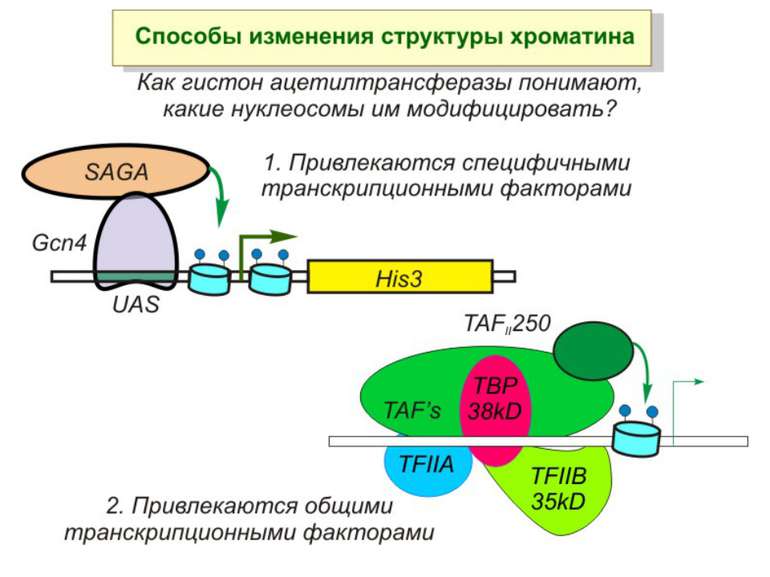
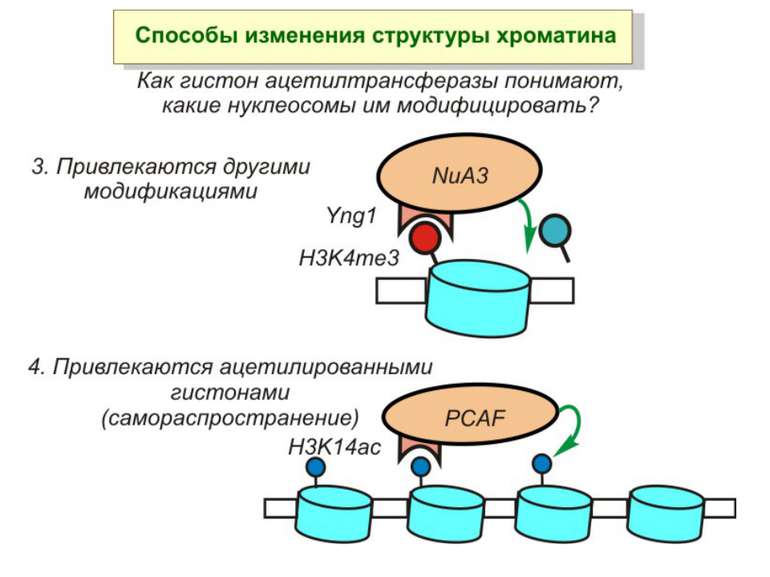
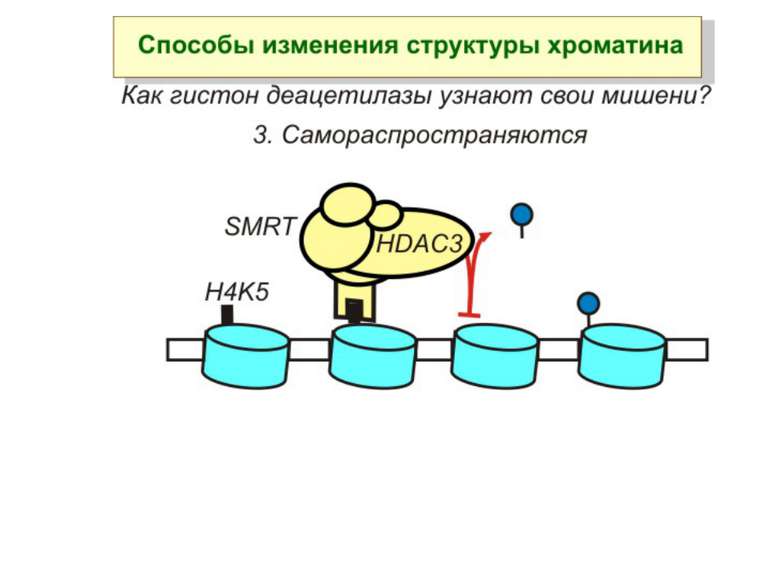
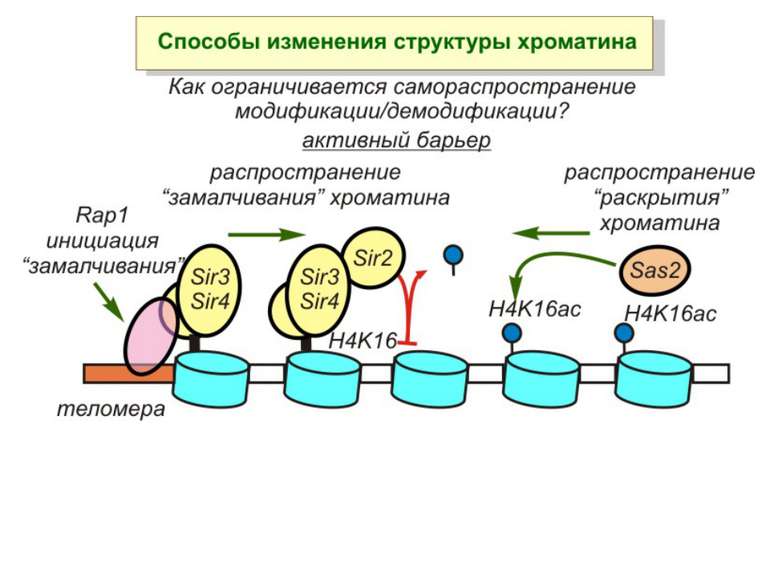
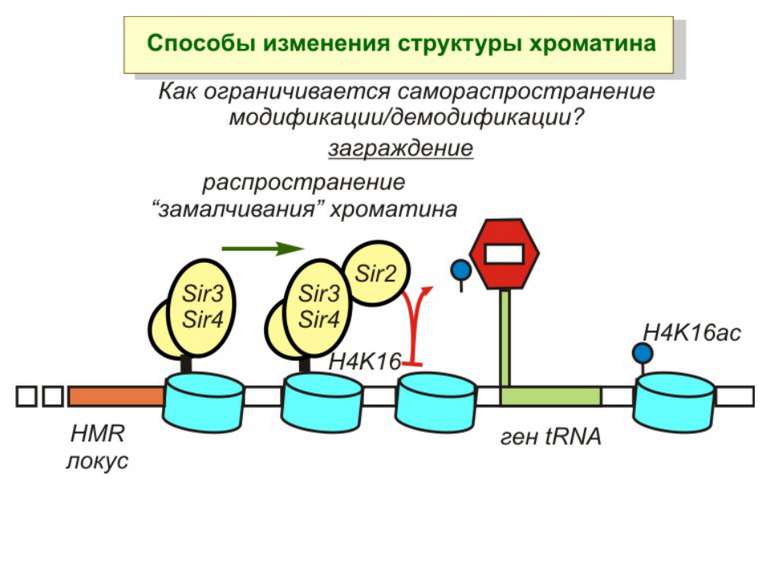
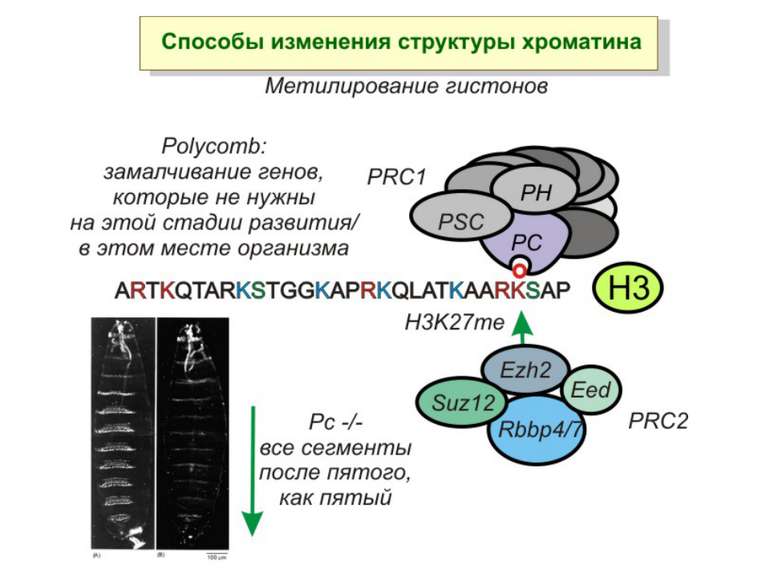
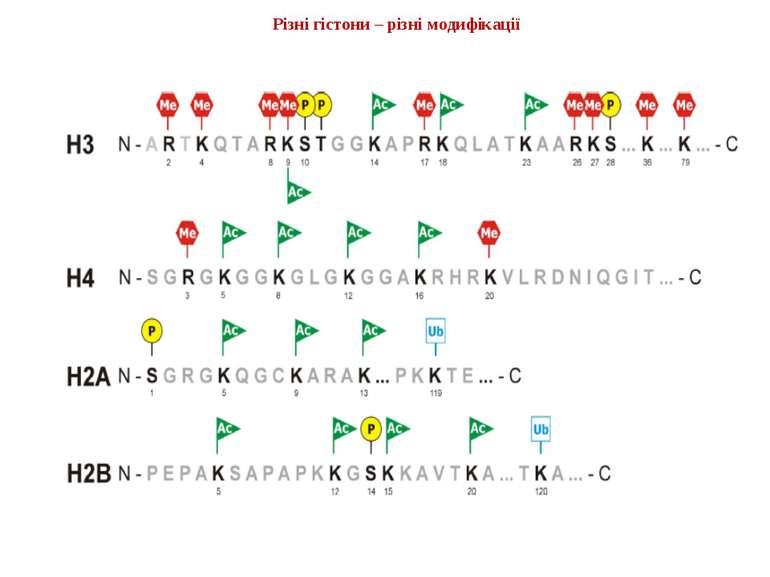
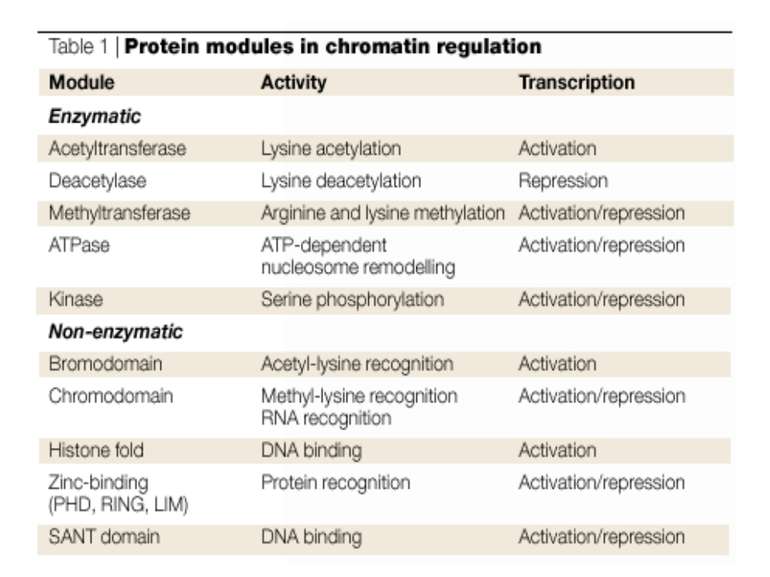
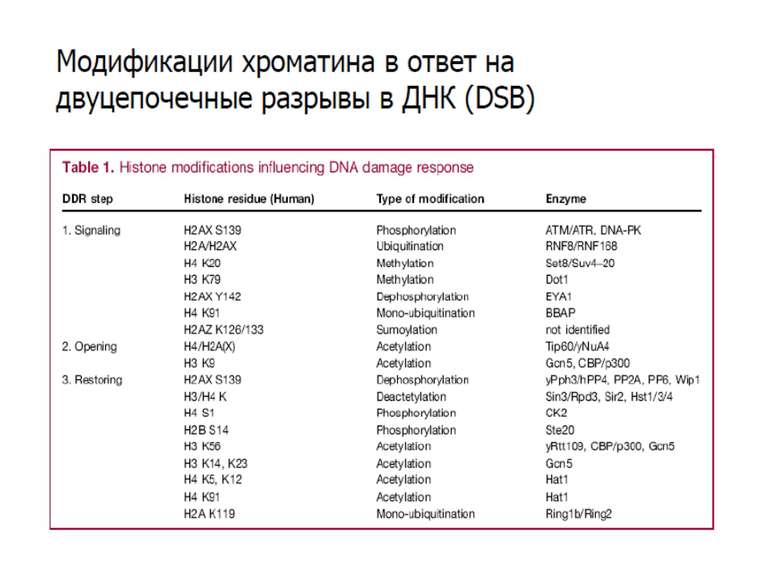
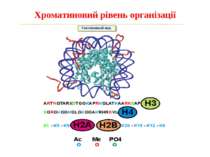
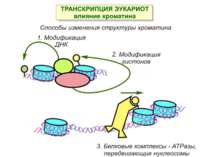
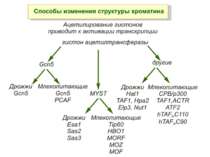
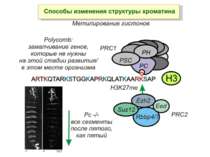
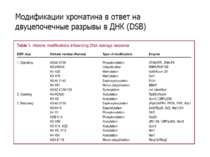
Способи зміни структури хроматину Модифікація гістонів високо консервативні лізини: гістона Н3 в положенні 9, 14, 18 і 23 гістона Н4 - 5, 8,12 і 16
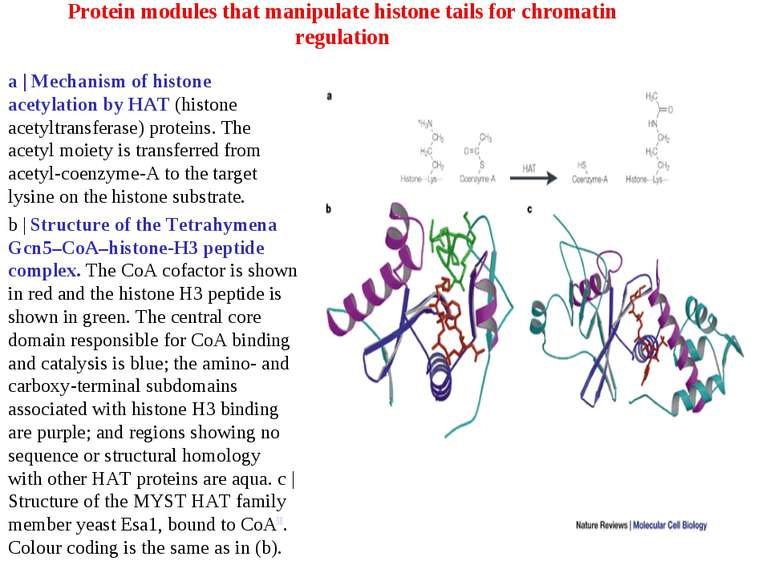
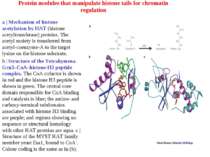
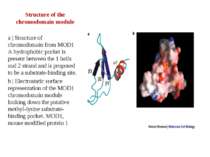
Protein modules that manipulate histone tails for chromatin regulation a | Mechanism of histone acetylation by HAT (histone acetyltransferase) proteins. The acetyl moiety is transferred from acetyl-coenzyme-A to the target lysine on the histone substrate. b | Structure of the Tetrahymena Gcn5–CoA–histone-H3 peptide complex. The CoA cofactor is shown in red and the histone H3 peptide is shown in green. The central core domain responsible for CoA binding and catalysis is blue; the amino- and carboxy-terminal subdomains associated with histone H3 binding are purple; and regions showing no sequence or structural homology with other HAT proteins are aqua. c | Structure of the MYST HAT family member yeast Esa1, bound to CoA58. Colour coding is the same as in (b).
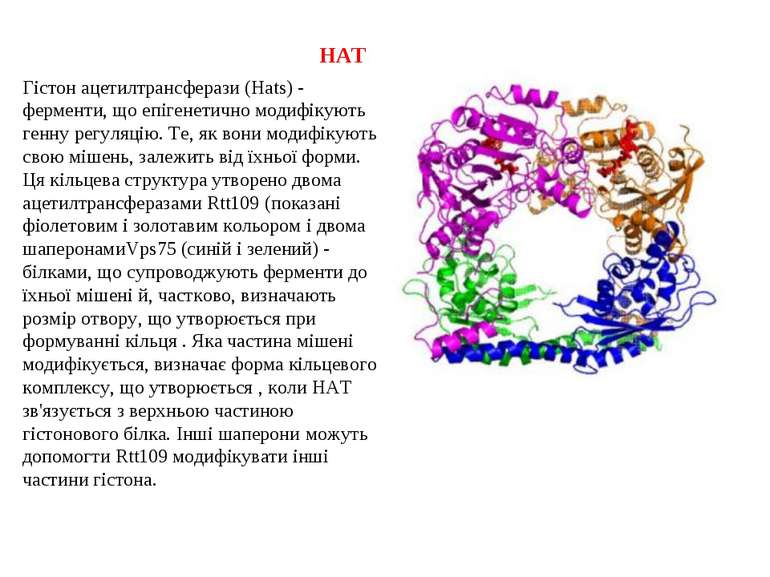
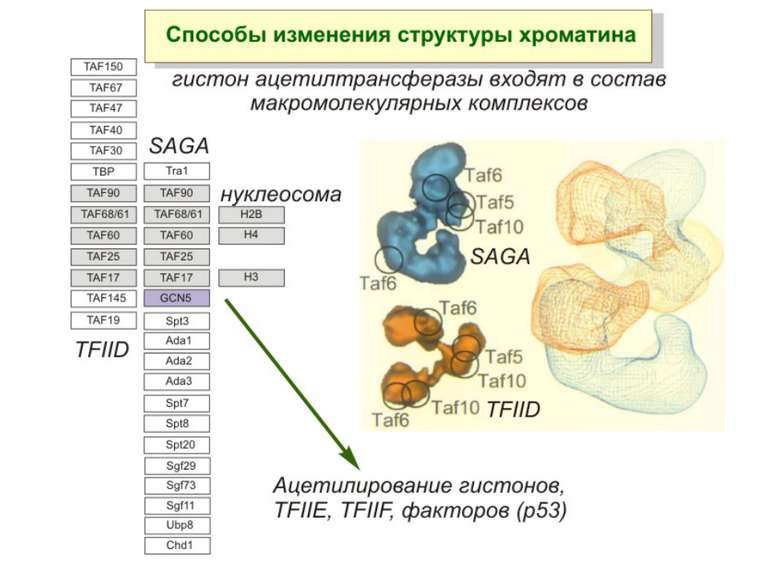
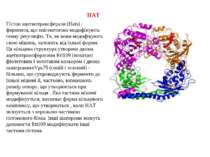
HAT Гістон ацетилтрансферази (Hats) - ферменти, що епігенетично модифікують генну регуляцію. Те, як вони модифікують свою мішень, залежить від їхньої форми. Ця кільцева структура утворено двома ацетилтрансферазами Rtt109 (показані фіолетовим і золотавим кольором і двома шаперонамиVps75 (синій і зелений) - білками, що супроводжують ферменти до їхньої мішені й, частково, визначають розмір отвору, що утворюється при формуванні кільця . Яка частина мішені модифікується, визначає форма кільцевого комплексу, що утворюється , коли HAT зв'язується з верхньою частиною гістонового білка. Інші шаперони можуть допомогти Rtt109 модифікувати інші частини гістона.
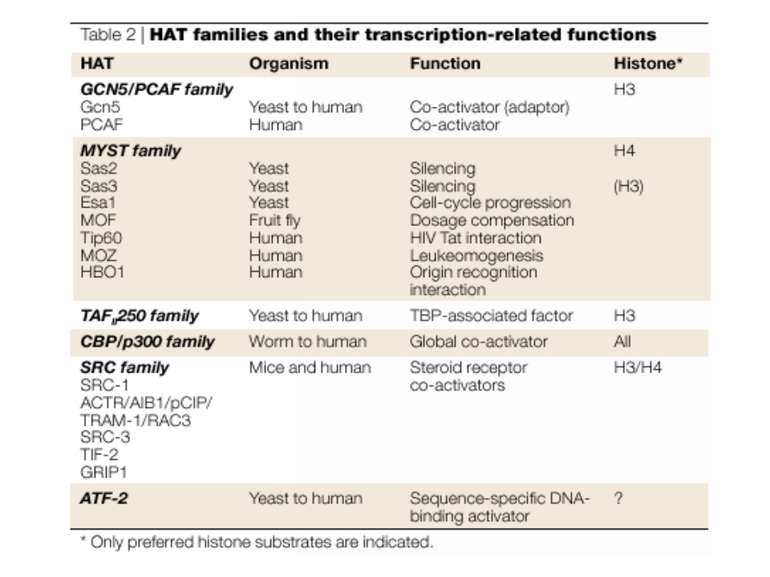
GNAT родина (Gcn5-related N-acetytransferases) включає білки, що беруть участь або зв’язані з ініціацією транскипції (Gcn5 і PCAF), елонгацією (Elp3), депозицією гістонів і теломерним мовчанням (Hat1)
Родина p300/CBP HAT Наявні спільні гомологічні послідовності з GNATs. HAT активність p300 і CBP необхідна для їх роли в трансактивації, вони асоціюють з ін. ацетилтрансферазами. P300/CBP - HAC, взаємодіють з енхансерами і асоціюють з РНК - полімеразою II
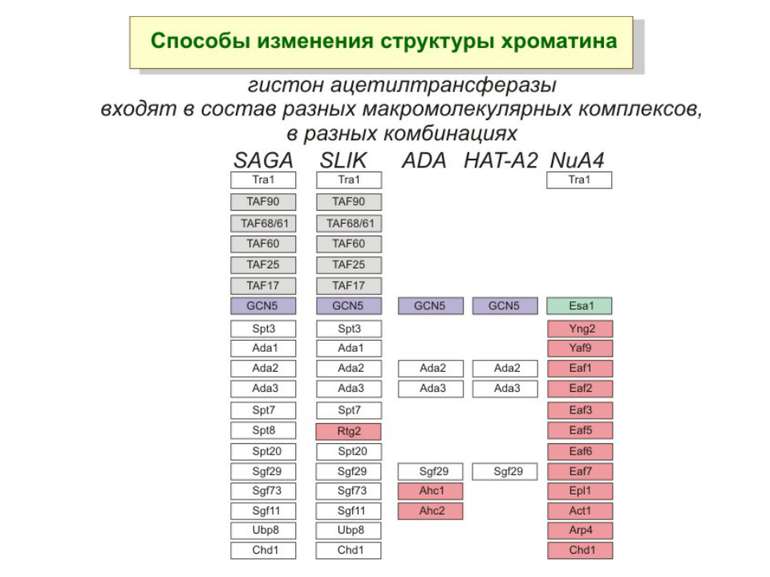
Родина MYST Включає членів MOZ (онкоген людини), Ybf2/Sas3, Sas2 і Tip60. Н.: гостра мієлома Участь в транскрипційній елонгації і реплікації та в мовчанні дріжджевих локусів HM типів спарювання. На відміну від SAGA, комплекс NuA3 не рекрутується дріжджевими промоторами за допомогою кислих активаторів.
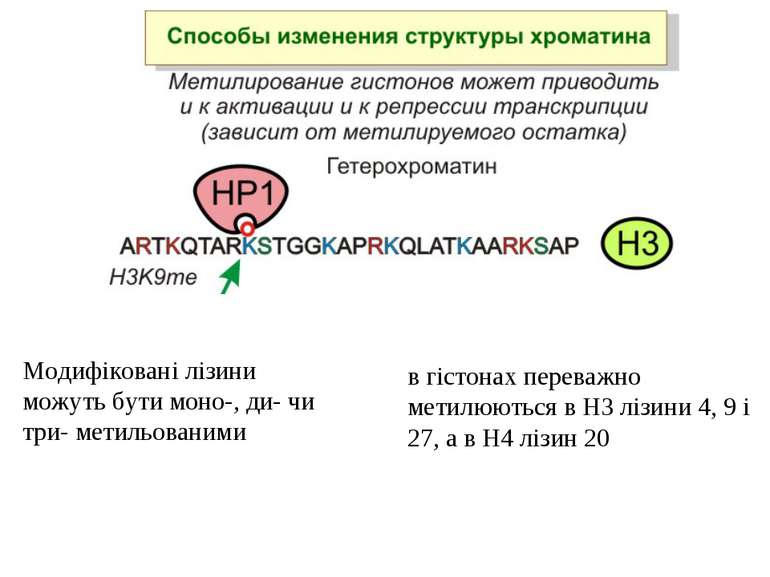
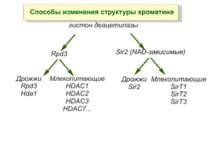
в гістонах переважно метилюються в H3 лізини 4, 9 і 27, а в H4 лізин 20 Модифіковані лізини можуть бути моно-, ди- чи три- метильованими
Фосфорилювання гістонів Тісно спряжене з ацетилюванням Н.: Ліз-9 Сер-10 Може відбувати у відповідь на пошкодження ДНК (ASQE-мотив Н2А)
NB !!! Метилювання і ацетилирювання Lys9 взаємовиключаючі. Метилювання Lys9 інгібує фосфорилювання Ser10, а не ацетилювання Lys14. Метилювання Lys9 корелює з мовчанням генів, тоді як фосфорилювання Ser10 і ацетилювання Lys9 корелює з транскрипційною активністю
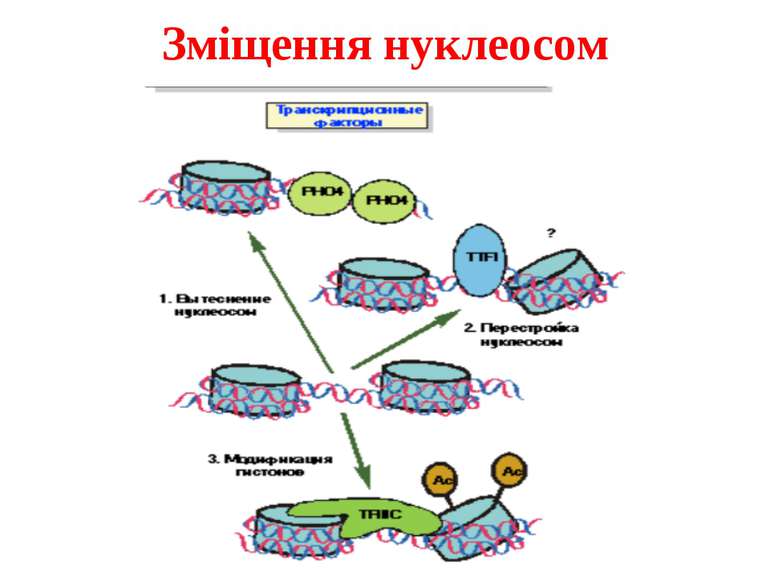
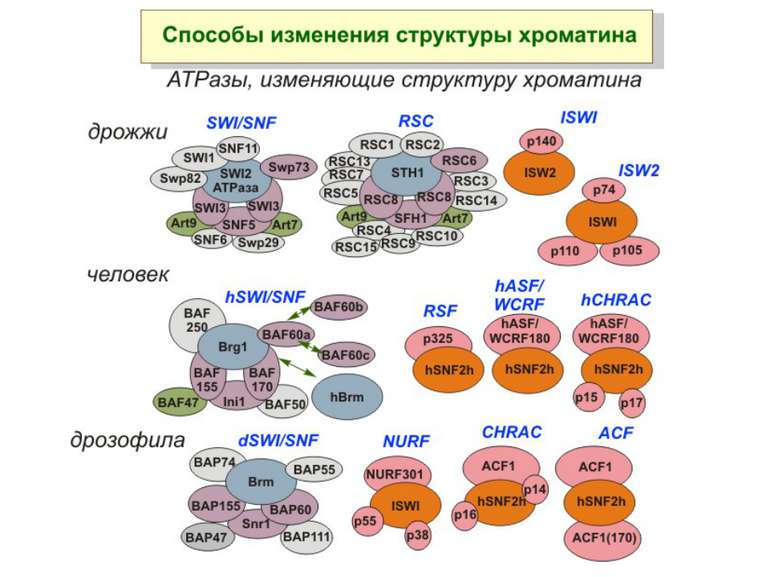
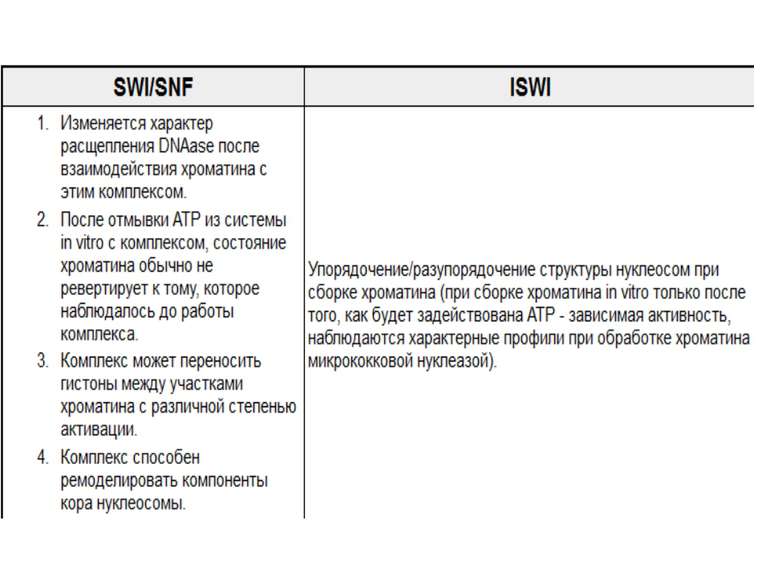
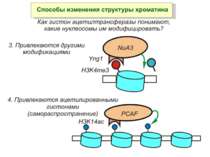
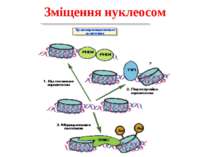
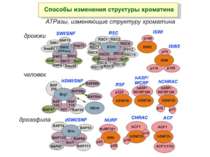
Комплекси, що містять гомологи SWI2 і ISWI відрізняються !!! SWI2 може використовувати як субстрат ненуклеосомну ДНК, і може виконувати складні перебудови ДНК-гістонового комплексу. Субстратом ISWI жуть бути лише нуклеосоми, а основною функцією – зсув нуклеосом по ДНК. Причому для ISWI необхідним є N-кінець гістону H4.
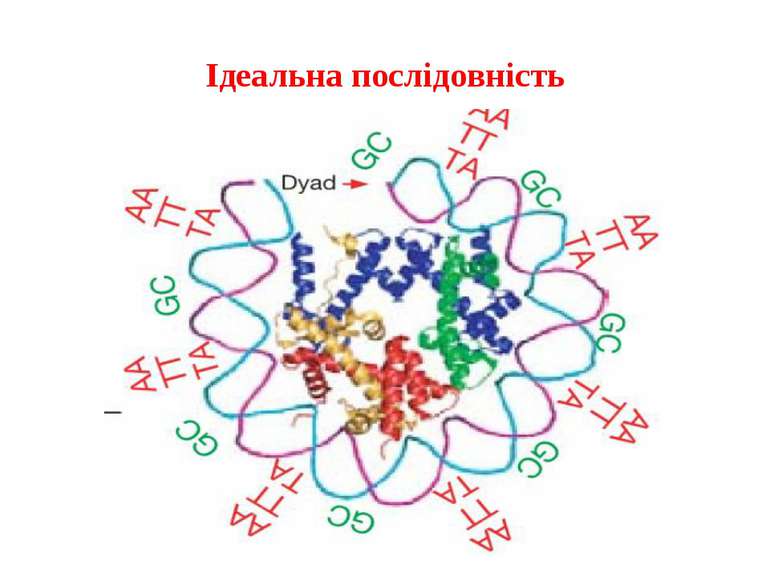
Де знаходяться гістони ? Послідовності ДНК можуть в 1000 раз відрізнятися по потенціалу зв'язувати нуклеосому Це пов'язане з анізотропною гнучкістю ДНК. Пари нуклеотидів AG/CT, CG/GC, GA/TC, GC/CG, AA/TT згинають ДНК Якщо підряд ідуть послідовності, що згинають ДНК в одну сторону (наприклад, TATA!), зв'язування нуклеосоми буде нестійким. Протилежно спрямований вигин ДНК із періодом, що відповідають 1 витку спиралі ДНК – максимально міцне зв'язування з нуклеосомою . Знаючи тільки первинну структуру ДНК можна передбачити точне положення більш 50% нуклеосом! («A genomic code for nucleosome positioning»)
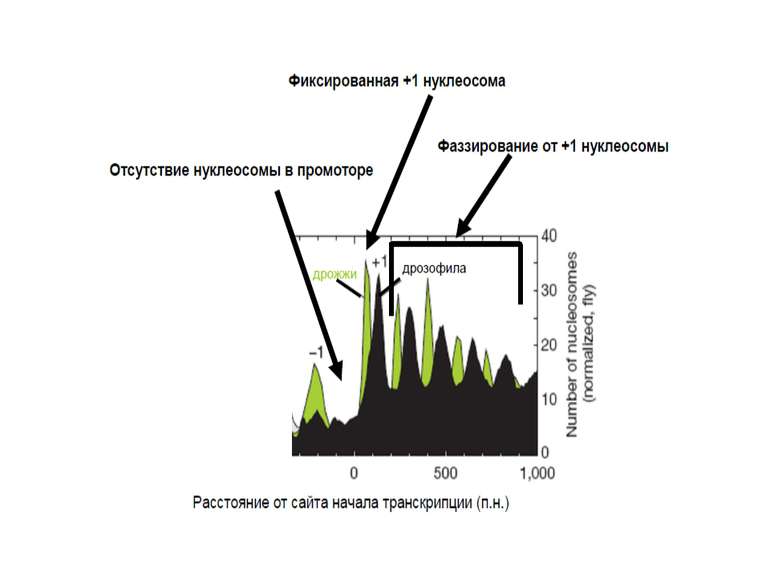
У геномі присутні Ділянки, вільні від нуклеосом сайти зв'язування транскрипційних факторів, регуляторних білків Ділянки, де положення нуклеосоми строго фіксоване +1 нуклеосома в генах (дріжджі – від +1 нуклеотиду, хребетні – від +60) Ділянки, у яких нуклеосомна укладка піддається регуляції білками АТФ-залежного ремоделінгу хроматину Ділянки, у яких спостерігається фазування нуклеосом, починаючи від фіксованої нуклеосоми
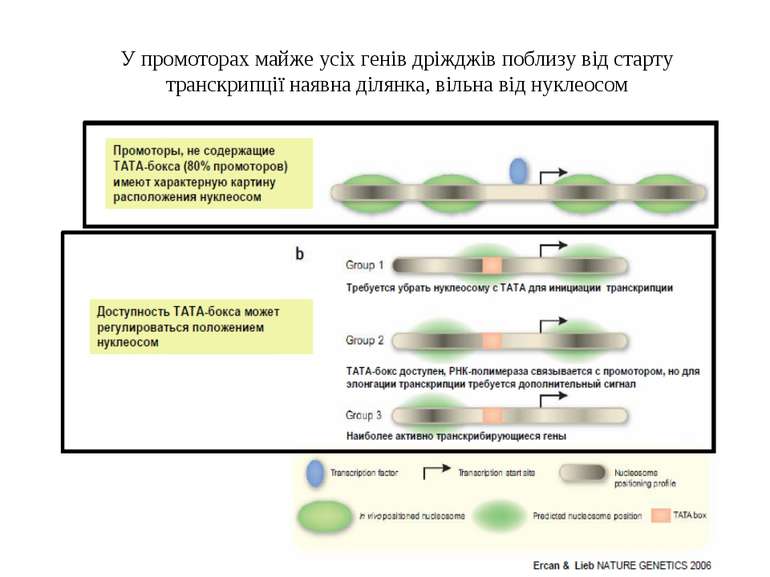
У промоторах майже усіх генів дріжджів поблизу від старту транскрипції наявна ділянка, вільна від нуклеосом
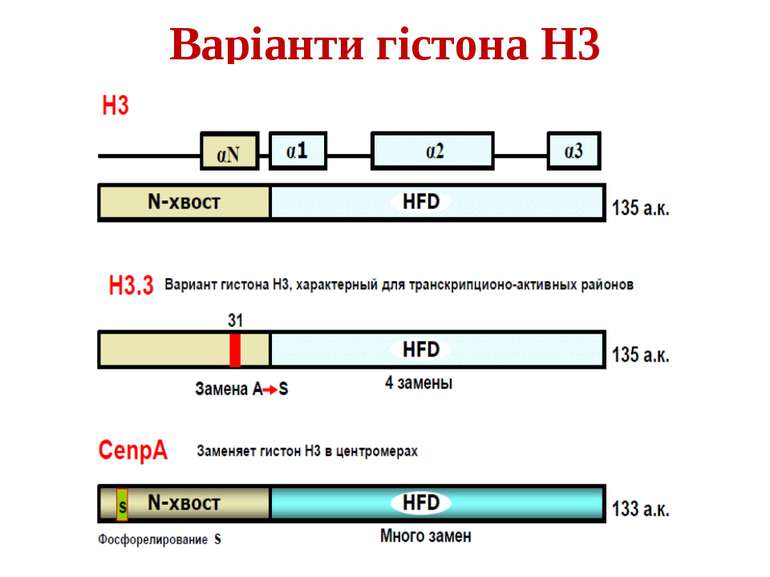
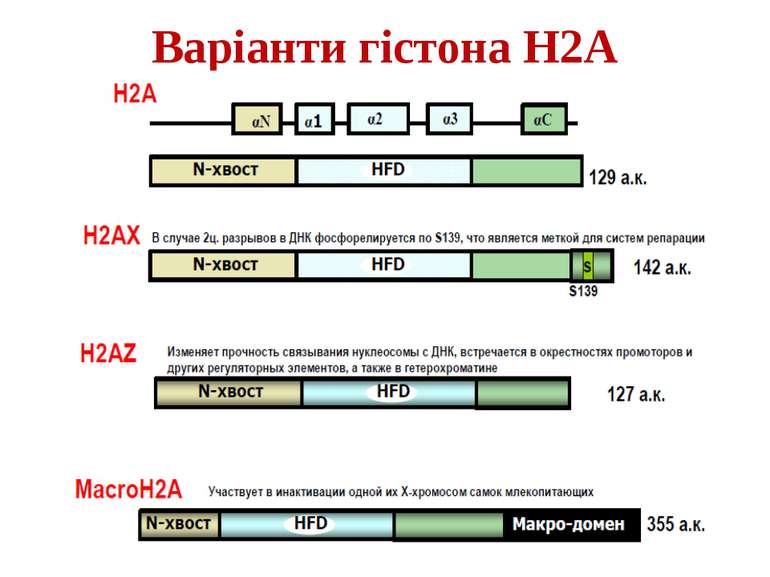
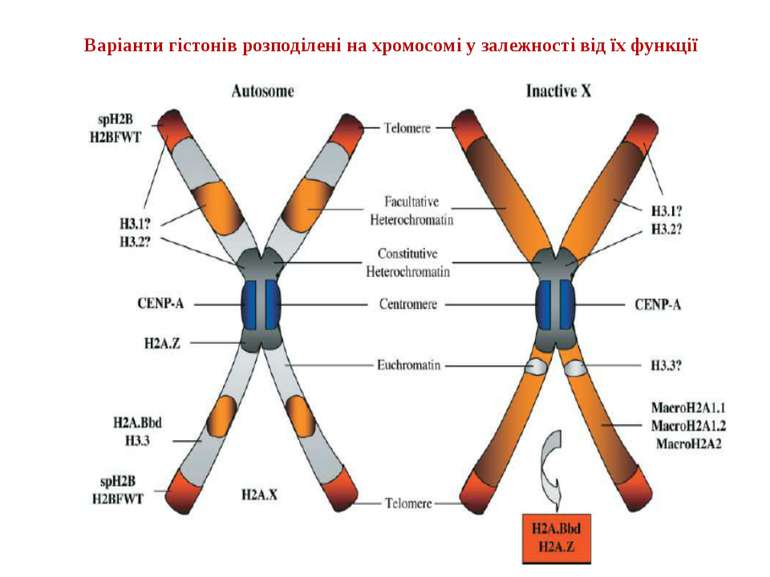
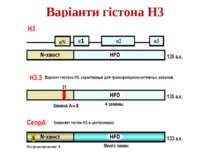
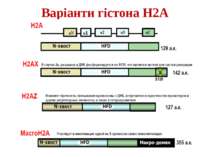
Стабільність зв'язування між ДНК і нуклеосомою може змінюватися при заміні основних варіантів гістонів на специфічні й у результаті ковалентних модифікацій гістонових хвостів Варіанти гістонів кодуються окремими генами. Вони гомологічні генам основного набору гістонів, але менш консервативні
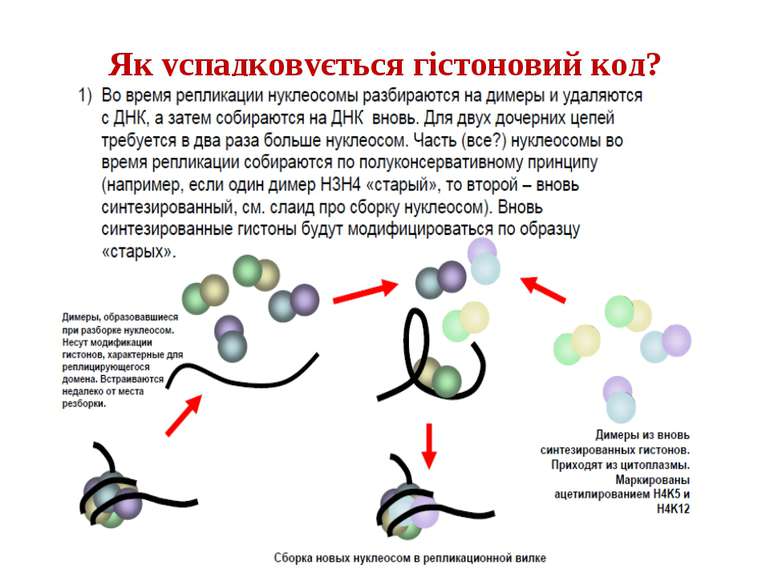
Посттрансляційні модифікації гістонів – “молекулярні мітки” Неепігенетичні Епігенетичні Успадковуються “гістоновий код”
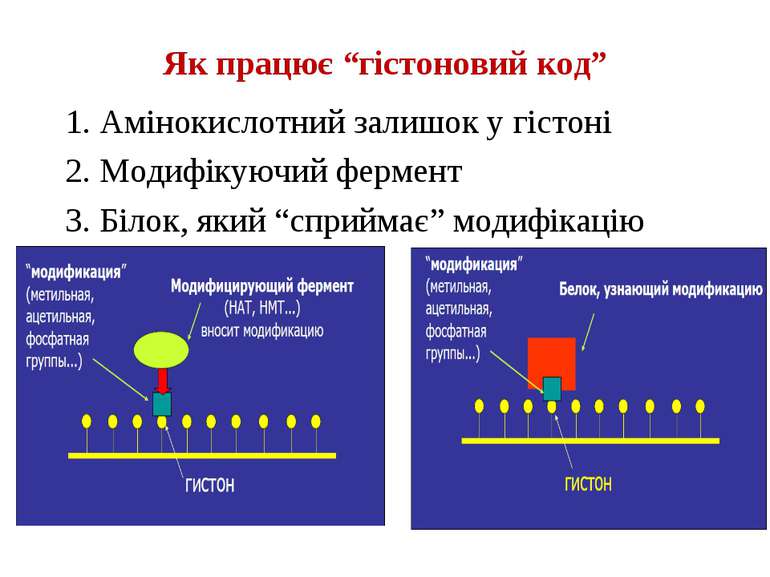
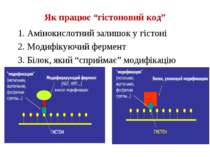
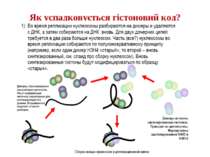
“Гістоновий код” різні паттерни модифікації гістонів (результат ацетилювання, фосфорилювання, метилювання і ін. пост-трансляційних модифікацій) діють послідовно або в комбінації для формування «гістонового коду», який зчитується за допомогою інших білків, щоб викликати певні низхідні транскрипційні події.
Як працює “гістоновий код” 1. Амінокислотний залишок у гістоні 2. Модифікуючий фермент 3. Білок, який “сприймає” модифікацію
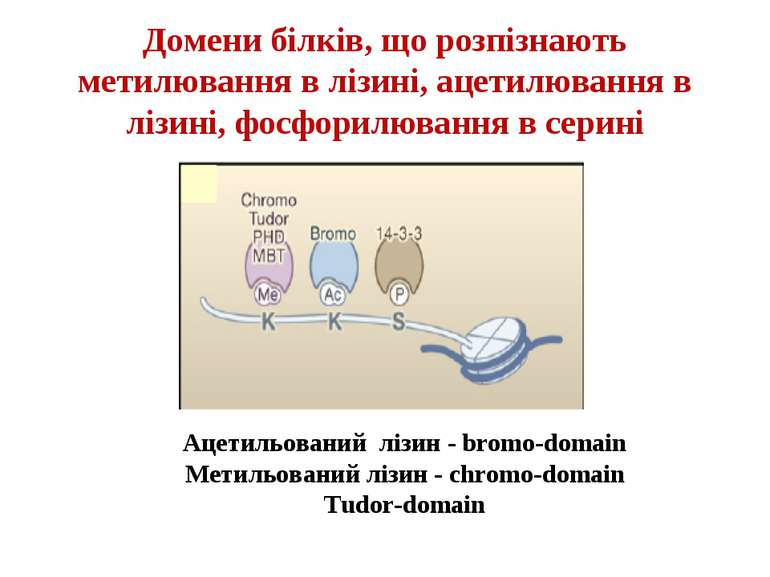
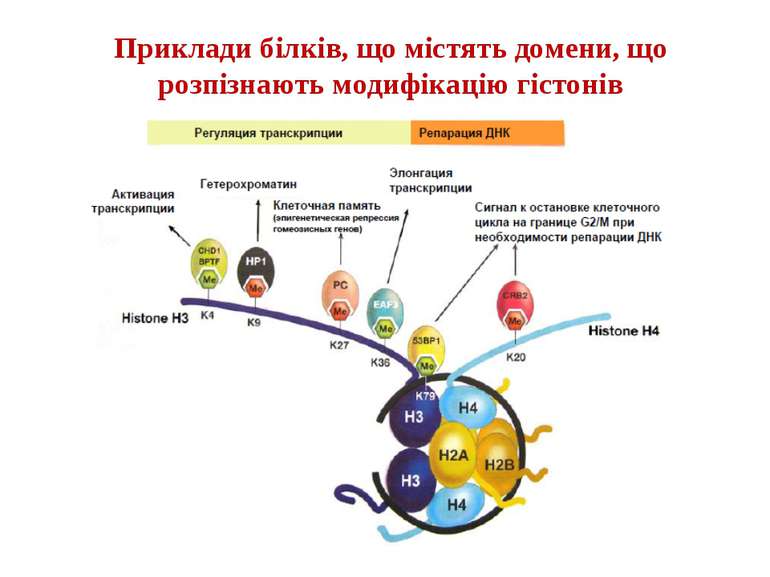
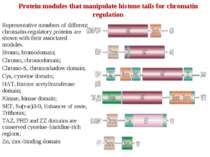
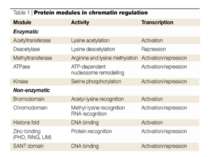
Домени білків, що розпізнають метилювання в лізині, ацетилювання в лізині, фосфорилювання в серині Ацетильований лізин - bromo-domain Метильований лізин - chromo-domain Tudor-domain
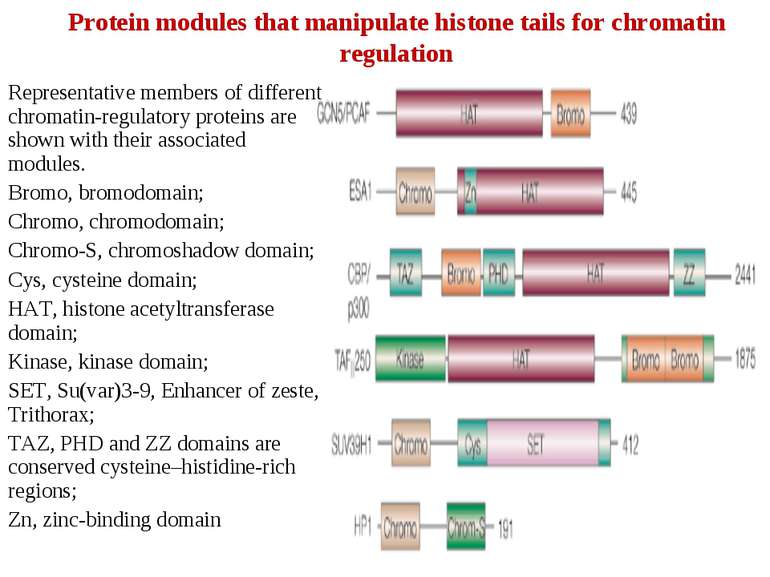
Protein modules that manipulate histone tails for chromatin regulation Representative members of different chromatin-regulatory proteins are shown with their associated modules. Bromo, bromodomain; Chromo, chromodomain; Chromo-S, chromoshadow domain; Cys, cysteine domain; HAT, histone acetyltransferase domain; Kinase, kinase domain; SET, Su(var)3-9, Enhancer of zeste, Trithorax; TAZ, PHD and ZZ domains are conserved cysteine–histidine-rich regions; Zn, zinc-binding domain
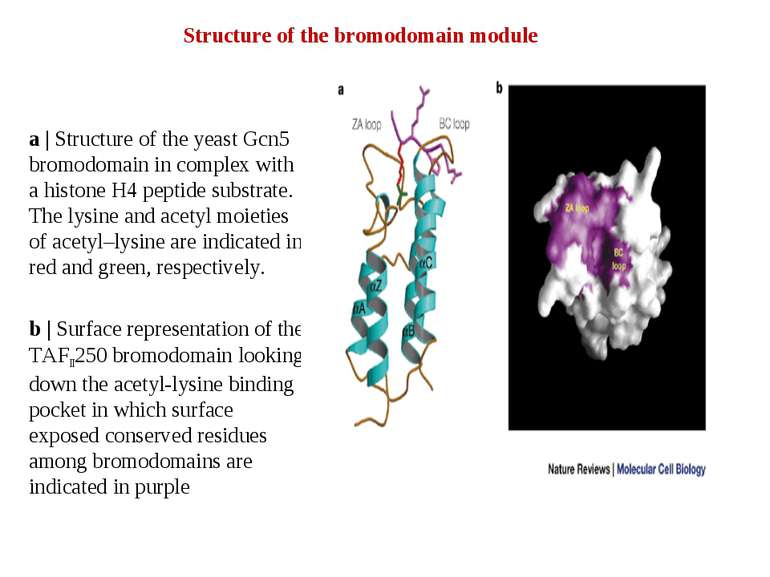
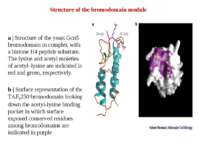
Structure of the bromodomain module a | Structure of the yeast Gcn5 bromodomain in complex with a histone H4 peptide substrate. The lysine and acetyl moieties of acetyl–lysine are indicated in red and green, respectively. b | Surface representation of the TAFII250 bromodomain looking down the acetyl-lysine binding pocket in which surface exposed conserved residues among bromodomains are indicated in purple
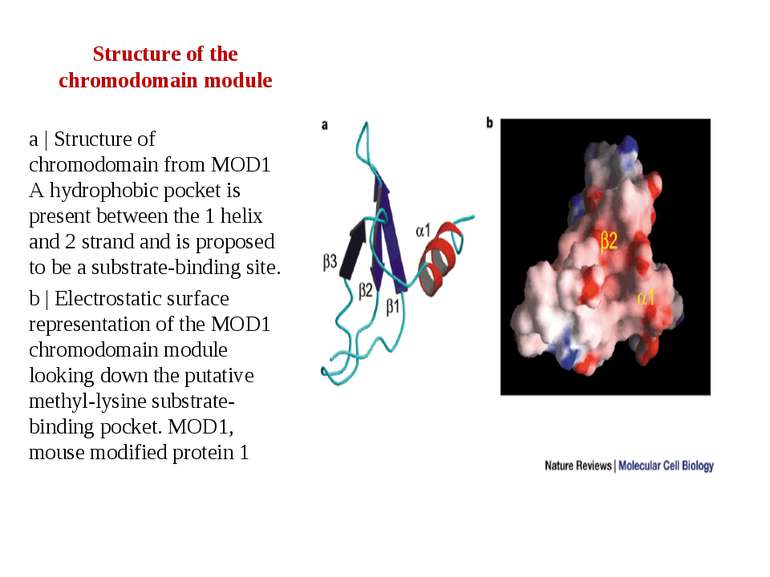
Structure of the chromodomain module a | Structure of chromodomain from MOD1 A hydrophobic pocket is present between the 1 helix and 2 strand and is proposed to be a substrate-binding site. b | Electrostatic surface representation of the MOD1 chromodomain module looking down the putative methyl-lysine substrate-binding pocket. MOD1, mouse modified protein 1
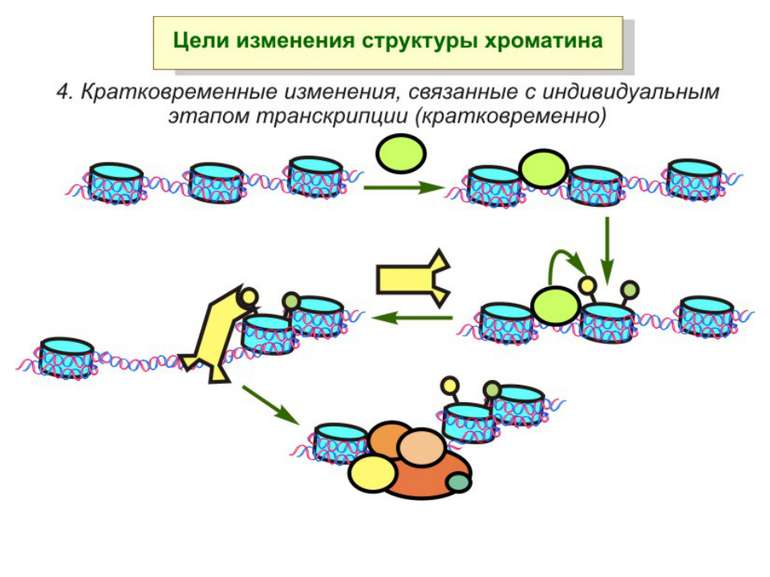
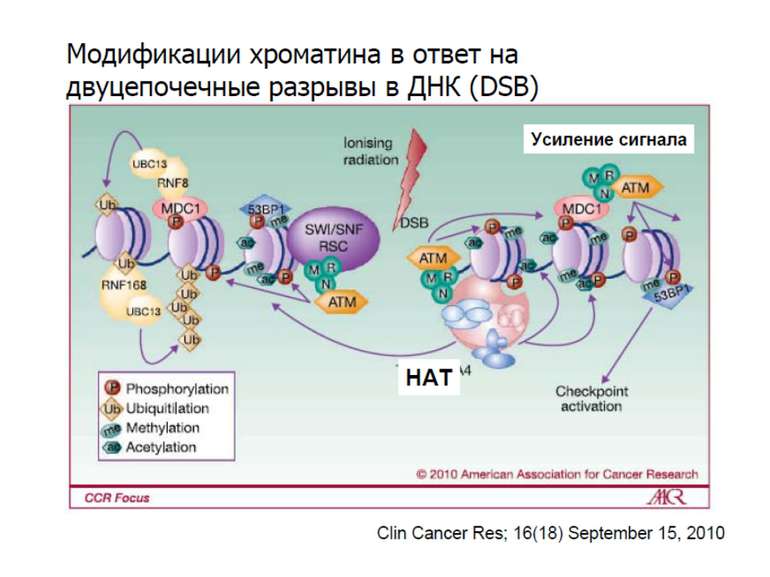
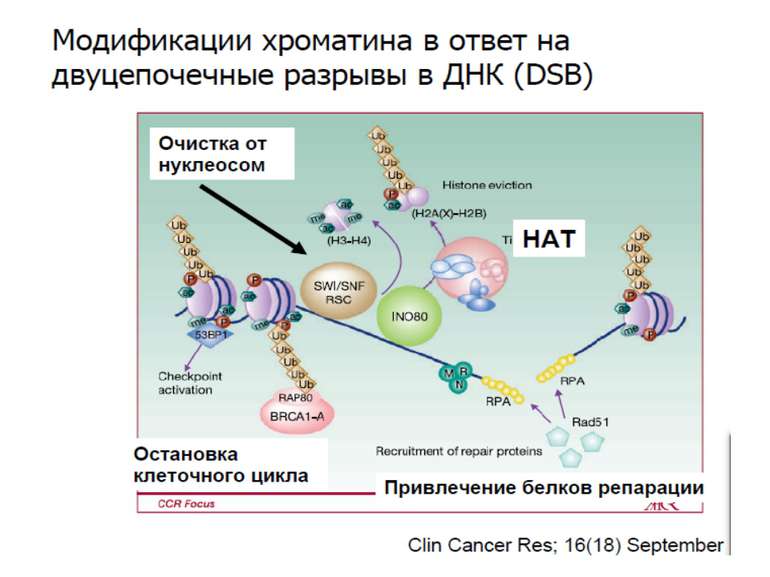
Після того, як відбулось ремоделювання структури хроматину HAC, SWI/SNF, ISWI комплексами, наступає час роботи РНК - полімерази II
Моделі транскрипції: На першому етапі із зарепресованим хроматином зв'язуються активатори, які сприяють роботі над усією послідовністю гена ремоделюючих комплексів, які, у свою чергу, роблять ДНК доступною для роботи РНК - полімерази II. Із зарепресованим хроматином зв'язуються активатори. Після цього уздовж послідовності гена проходить "первинна" РНК - полімераза, яка тягне на собі все необхідне для ремоделювання хроматину. При цьому полімераза транскрибує ген. Є експериментальні дані, що вказують на те, що первинний транскрипт відрізняється від наступних транскриптів з даного гена в даному активному стані. Слідом за "первинною" полімеразою починає працювати РНК - полімераза II, яка рухається вже по ремодельованому хроматину уздовж послідовності гена.
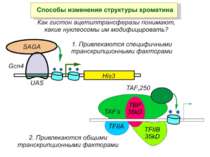
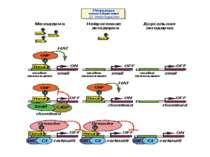
Приклади дотранскрипційних подій з дріжджовим промотором HO спочатку зв'язується SWI/SNF, потім SAGA. Після цих хроматин-модифікуючих комплексів зв'язується активатор, що «притягує» полімеразу за SRB/медіаторний комплекс ( що сидить на CTD домені РНК полімерази II). Викликана вірусом активація промотору інтерферону. Спочатку діє гістон-ацетилтрансфераза GCN5, потім гістон-ацетилтрансфераза CBP і відбувається посадка РНК-полімерази II. CBP приваблює SWI/SNF, який відсуває нуклеосому від TATA послідовності й, таким чином, звільняє місце для посадки TFIID. a 1-антитрипсиновий промотор. У неактивному стані з ним зв'язаний активатор HNF-1a, TFIIB і TBP. При активації спочатку збирається повний TFIID і приєднується пролімераза, потім – гістонацетилтрансферази CBP і PCAF. Після гіперацетилювання гістонів у районі промотору, приєднується HNF4 і SWI/SNF. Після цього відбувається ініціація.
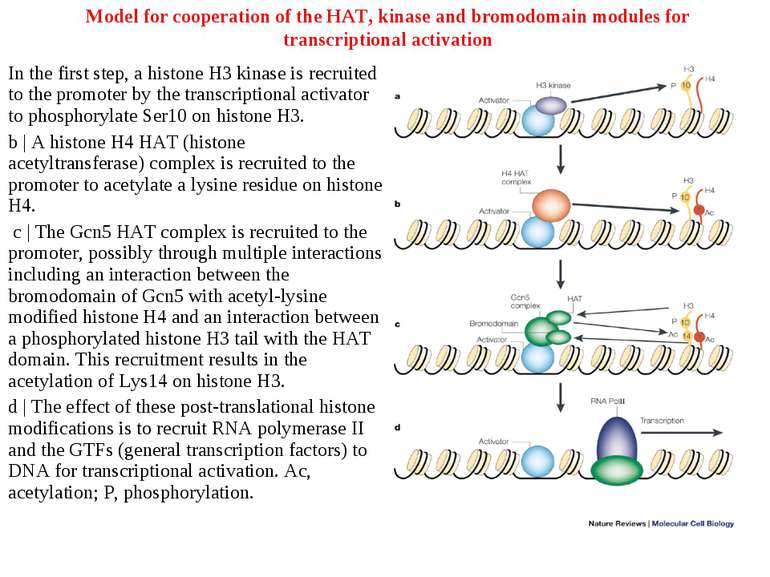
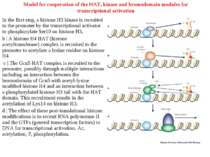
Model for cooperation of the HAT, kinase and bromodomain modules for transcriptional activation In the first step, a histone H3 kinase is recruited to the promoter by the transcriptional activator to phosphorylate Ser10 on histone H3. b | A histone H4 HAT (histone acetyltransferase) complex is recruited to the promoter to acetylate a lysine residue on histone H4. c | The Gcn5 HAT complex is recruited to the promoter, possibly through multiple interactions including an interaction between the bromodomain of Gcn5 with acetyl-lysine modified histone H4 and an interaction between a phosphorylated histone H3 tail with the HAT domain. This recruitment results in the acetylation of Lys14 on histone H3. d | The effect of these post-translational histone modifications is to recruit RNA polymerase II and the GTFs (general transcription factors) to DNA for transcriptional activation. Ac, acetylation; P, phosphorylation.
Схожі презентації
Категорії